Emersione di SARS-CoV-2 B.1.1.7 Lignaggio—
Stati Uniti, 29 dicembre 2020–12 gennaio 2021
Estate E. Galloway, PhD 1;Prabasaj Paolo, PhD 1;Duncan R. MacCannell, PhD 2;Michael A. Johansson, PhD 1;
John T. Brooks, MD 1;Adam MacNeil, PhD 1;Rachel B. Slayton, PhD 1;Suxiang Tong, PhD 1;Benjamin J. Seta, PhD 1;Gregory L. Armstrong, MD 2;
Matthew Biggerstaff, ScD 1;Vivien G. Dugan, dottore di ricerca
Il 15 gennaio 2021, questo rapporto è stato pubblicato come MMWRRilascio anticipato sul sito web di MMWR (https://www.cdc.gov/mmwr).
Il 14 dicembre 2020, il Regno Unito ha riferitouna variante preoccupante di SARS-CoV-2 (VOC), lignaggio B.1.1.7,indicato anche come VOC 202012/01 o 20I/501Y.V1.* IlSi stima che la variante B.1.1.7 sia emersa a settembre2020 ed è diventato rapidamente il dominante in circolazioneVariante SARS-CoV-2 in Inghilterra (1).B.1.1.7 è statorilevato in oltre 30 paesi, inclusi gli Stati Uniti.COMEdel 13 gennaio 2021, circa 76 casi di B.1.1.7 hannostato rilevato in 12 stati degli Stati Uniti.Molteplici linee di provaindicano che B.1.1.7 è trasmesso in modo più efficiente di quanto lo sianoaltre varianti SARS-CoV-2 (1–3).La traiettoria modellata diquesta variante negli Stati Uniti mostra una rapida crescita all'inizio del 2021,diventando la variante predominante a marzo.È aumentatoLa trasmissione di SARS-CoV-2 potrebbe minacciare l'assistenza sanitaria tesarisorse, richiedono un'implementazione estesa e più rigorosadelle strategie di sanità pubblica (4) e aumentare la percentuale diimmunità della popolazione necessaria per il controllo della pandemia.Prendendole misure per ridurre la trasmissione ora possono diminuire il potenzialeimpatto di B.1.1.7 e consentire un tempo critico per aumentare la vaccinazionecopertura zione.Collettivamente, sorveglianza genomica potenziatacombinato con la continua conformità con il pubblico effettivomisure sanitarie, compresa la vaccinazione, il distanziamento fisico,uso di mascherine, igiene delle mani, isolamento e quarantena, lo farannoessenziale per limitare la diffusione del virus SARS-CoV-2che causa la malattia da coronavirus 2019 (COVID-19).Strategicotest su persone senza sintomi ma a più alto rischio diinfezione, come quelli esposti a SARS-CoV-2 o che hannofrequente inevitabile contatto con il pubblico, fornisce un altroopportunità di limitare la diffusione in corso.
Sorveglianza genomica globale e condivisione rapida open-sourceing delle sequenze del genoma virale hanno facilitato quasi in tempo realerilevamento, confronto e monitoraggio dell'evoluzione della SARS-CoV-2varianti che possono informare gli sforzi di sanità pubblica per controllare ilpandemia.Mentre alcune mutazioni nel genoma viraleemergere e poi retrocedere, altri potrebbero conferire un vantaggio selettivotage alla variante, compresa la trasmissibilità migliorata, in modo cheuna tale variante può rapidamente dominare altre varianti circolanti.
All'inizio della pandemia, varianti di SARS-CoV-2 contenentila mutazione D614G nella proteina spike (S) che aumental'avidità di legame al recettore divenne rapidamente dominante in moltiregioni geografiche (5,6).Alla fine dell'autunno 2020, più paesi hanno segnalato il rilevamentoVarianti SARS-CoV-2 che si diffondono in modo più efficiente.Inoltrealla variante B.1.1.7, varianti degne di nota includono la B.1.351lignaggio rilevato per la prima volta in Sud Africa e recentemente identificatoB.1.1.28 sottoclade (rinominata“P.1") rilevato in quattro viaggiatoridal Brasile durante la proiezione di routine all'Haneda (Tokyo)aeroporto.§ Queste varianti portano una costellazione di mutazioni genetichezioni, incluso nel dominio di legame del recettore della proteina S,che è essenziale per il legame alla cellula ospite angiotensina-conversione del recettore dell'enzima-2 (ACE-2) per facilitare il virusiscrizione.L'evidenza suggerisce che altre mutazioni trovate in questile varianti potrebbero conferire non solo una maggiore trasmissibilità mapotrebbe anche influire sulle prestazioni di alcune diagnostiche in tempo realetrascrizione inversa–reazione a catena della polimerasi (RT-PCR)saggi¶ e ridurre la suscettibilità agli anticorpi neutralizzanti(2,3,5–10).Un recente case report ha documentato il primo caso diReinfezione da SARS-CoV-2 in Brasile con una variante SARS-CoV-2che conteneva la mutazione E484K,** che è stata dimostrataper ridurre la neutralizzazione da sieri di convalescenza e monoclonalianticorpi (9,10).
Questo rapporto si concentra sull'emergere della variante B.1.1.7negli Stati Uniti.A partire dal 12 gennaio 2021, né ilB.1.351 né le varianti P.1 sono state rilevate nelStati Uniti.Per informazioni sull'emergente SARS-CoV-2varianti di preoccupazione, CDC mantiene una pagina web dedicata afornire informazioni sulle varianti emergenti di SARS-CoV-2.††
B.1.1.7 lignaggio (20I/501Y.V1)
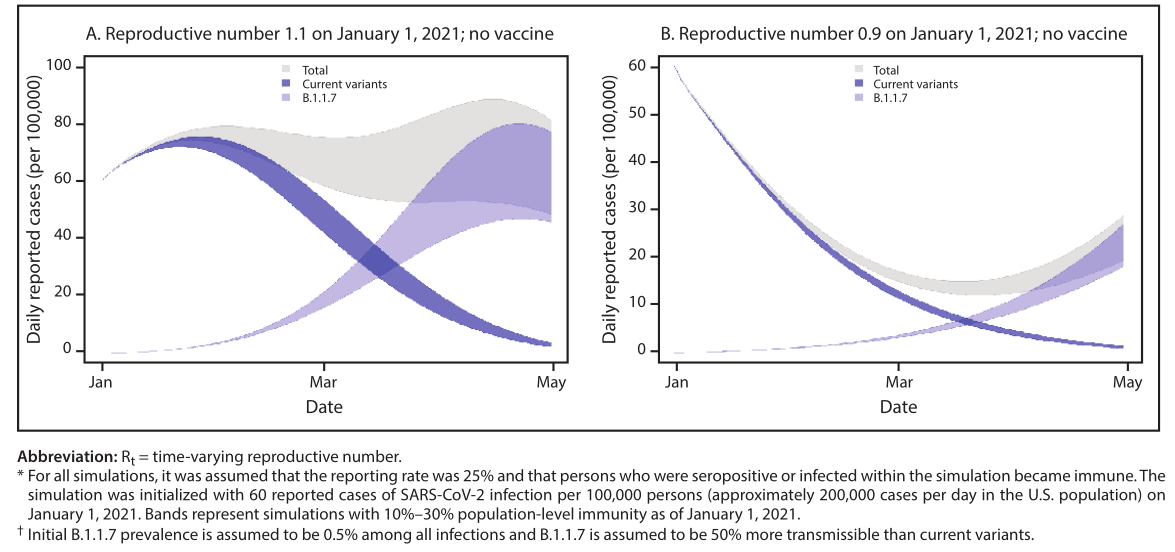
La variante B.1.1.7 porta una mutazione nella proteina S(N501Y) che influisce sulla conformazione del legame al recettoredominio.Questa variante ha altre 13 mutazioni che definiscono il lignaggio B.1.1.7 (Tabella), molte delle quali sono nella proteina S,inclusa una cancellazione alle posizioni 69 e 70 (del69–70) chesi è evoluto spontaneamente in altre varianti di SARS-CoV-2 ed èipotizzato per aumentare la trasmissibilità (2,7).La cancellazionenelle posizioni 69 e 70 provoca il fallimento del bersaglio del gene S (SGTF)in almeno una RT-PCR–test diagnostico basato (cioè, con ilSaggio ThermoFisher Taq Path COVID-19, B.1.1.7 variant e altre varianti con il del69–70 producono un negativorisultato per il bersaglio del gene S e un risultato positivo per gli altri dueobiettivi);SGTF ha svolto il ruolo di proxy nel Regno Unitoper identificare i casi B.1.1.7 (1).Molteplici linee di evidenza indicano che B.1.1.7 è di piùtrasmesso in modo efficiente rispetto ad altri SARS-CoV-2varianti in circolazione nel Regno Unito.Regioni del Regno Unito conuna percentuale maggiore di sequenze B.1.1.7 ha avuto un'epidemia più rapidacrescita rispetto ad altre aree, le diagnosi di SGTF sono aumentatepiù veloce rispetto alle diagnosi non SGTF nelle stesse aree, e apercentuale più elevata di contatti è stata infettata da pazienti indicecon infezioni B.1.1.7 rispetto ai pazienti indice con infezionealtre varianti (1,3).La variante B.1.1.7 ha il potenziale per aumentare la panoramica degli Stati Unitidemica nei prossimi mesi.Per illustrare questo effetto,è stato sviluppato un semplice modello compartimentale a due varianti.L'attuale prevalenza negli Stati Uniti di B.1.1.7 tra tutti i circolantivirus è sconosciuto ma si pensa che sia <0,5% in base alnumero limitato di casi rilevati e dati SGTF (8).Permodello, le assunzioni iniziali includevano una prevalenza B.1.1.7dello 0,5% tra tutte le infezioni, immunità SARS-CoV-2 daprecedente infezione del 10%–30%, un riproduttore variabile nel temponumero (R t ) di 1.1 (trasmissione mitigata ma in aumento)o 0,9 (diminuzione della trasmissione) per le varianti attuali e un'incidenza riportata di 60 casi per 100.000 persone al giorno il1 gennaio 2021. Queste ipotesi non rappresentano esattamentequalsiasi singola località degli Stati Uniti, ma piuttosto indica una generalizzazione dicondizioni comuni in tutto il paese.La variazione di R t finitatempo derivante dall'immunità acquisita e dall'aumento della prevalenza di B.1.1.7, è stata modellata, assumendo B.1.1.7 R tessere una costante 1,5 volte l'R t delle varianti attuali, in base astime iniziali dal Regno Unito (1,3).Successivamente, è stato modellato il potenziale impatto della vaccinazioneipotizzando che siano state somministrate 1 milione di dosi di vaccino pergiorno a partire dal 1 gennaio 2021 e quell'immunità del 95%.è stato raggiunto 14 giorni dopo aver ricevuto 2 dosi.Nello specifico,immunità contro l'infezione con le varianti attuali o ilSi è ipotizzata la variante B.1.1.7, sebbene l'efficacia ela durata della protezione contro le infezioni rimane incerta,perché questi non erano l'endpoint primario degli studi cliniciper i primi vacciniIn questo modello, la prevalenza di B.1.1.7 è inizialmente bassa, ma perchéè più trasmissibile delle attuali varianti, mostrarapida crescita all'inizio del 2021, diventando la varietà predominanteformica in marzo (figura 1).Se trasmissione di correntevarianti è in aumento (iniziale R t = 1.1) o lentamente in diminuzione(iniziale R t = 0,9) a gennaio, B.1.1.7 determina un cambiamento sostanzialenella traiettoria di trasmissione e una nuova fase di esponenzialecrescita.Con la vaccinazione che protegge dalle infezioni, ille prime traiettorie epidemiche non cambiano e B.1.1.7 si diffondesi verifica ancora (Figura 2).Tuttavia, dopo che B.1.1.7 diventa ilvariante dominante, la sua trasmissione è stata sostanzialmente ridotta.L'effetto della vaccinazione sulla riduzione della trasmissione nel vicinotermine era massimo nello scenario in cui si trovava la trasmissionegià decrescente (R iniziale t = 0,9) (Figura 2).I primi sforzi chepuò limitare la diffusione della variante B.1.1.7, come universale euna maggiore conformità con le strategie di mitigazione della salute pubblica,consentirà più tempo alla vaccinazione in corso per ottenere risultati più elevatiimmunità a livello di popolazione.
Discussione
Attualmente, non vi è alcuna differenza nota nei risultati cliniciassociato alle varianti SARS-CoV-2 descritte;Tuttavia,un tasso di trasmissione più elevato porterà a più casi, in aumentoil numero complessivo di persone che necessitano di assistenza clinica, exacerscaricando l'onere su un sistema sanitario già teso,e provocando più morti.Sorveglianza genomica continuaidentificare i casi B.1.1.7, così come l'emergere di altrivarianti di preoccupazione negli Stati Uniti, è importante per ilRisposta di sanità pubblica COVID-19.Considerando che i risultati SGTFpuò aiutare a identificare potenziali casi B.1.1.7 che possono essere confermatimediante sequenziamento, identificando le varianti prioritarie che non presentanoSGTF si basa esclusivamente sulla sorveglianza basata sulla sequenza.
|
Designazione variante | Prima identificazione | Mutazioni caratteristiche (proteina: mutazione) | N. di casi confermati con sequenza corrente | N. di paesi con sequenze | ||
| Posizione | Data | stati Uniti | In tutto il mondo | |||
| B.1.1.7 (20I/501Y.V1) | Regno Unito | settembre 2020 | ORF1ab: T1001I, A1708D, I2230T, del3675–3677 SGF S: del69–70 HV, del144 Y, N501Y, A570D, D614G, P681H, T761I, S982A, D1118H ORF8: Q27stop, R52I, Y73C N: D3L, S235F | 76 | 15.369 | 36 |
| B.1.351 (20H/501Y.V2) | Sud Africa | ottobre 2020 | ORF1ab: K1655N E: P71L N: T205I S:K417N, E484K, N501Y, D614G, A701V | 0 | 415 | 13
|
| P.1 (20J/501Y.V3 | Brasile e Giappone | gennaio 2021 | ORF1ab: F681L, I760T, S1188L, K1795Q, del3675–3677 SGF, E5662D S: L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, T1027I ORF3a: C174G ORF8: E92K ORF9: Q77E ORF14: V49L N: P80R | 0 | 35 | 2
|
Abbreviazioni: del = cancellazione;E = proteina dell'involucro;N = proteina nucleocapside;ORF = cornice di lettura aperta;S = proteina spike.
L'esperienza nel Regno Unito ei modelli B.1.1.7presentati in questa relazione illustrano l'impatto un più contagiosovariante può avere sul numero di casi in una popolazione.ILmaggiore trasmissibilità di questa variante richiede ancora di piùrigorosa attuazione combinata di vaccinazione e mitigazionemisure di sicurezza (p. es., distanziamento, mascheramento e igiene delle mani)per controllare la diffusione di SARS-CoV-2.Queste misure sarannopiù efficaci se vengono istituiti prima piuttosto che dopoper rallentare la diffusione iniziale della variante B.1.1.7.Sforzi perpreparare il sistema sanitario per ulteriori ondate di casigarantito.Una maggiore trasmissibilità significa anche quella superioredeve essere raggiunta una copertura vaccinale superiore a quella previstaraggiungere lo stesso livello di controllo delle malattie per proteggere il pubblicorispetto alle varianti meno trasmissibili.In collaborazione con istituzioni accademiche, industriali, statali, territoriali,partner tribali e locali, CDC e altre agenzie federalistanno coordinando e potenziando la sorveglianza genomica esforzi di caratterizzazione del virus negli Stati Uniti.Centro per la prevenzione e il controllo delle malattiecoordina gli sforzi di sequenziamento degli Stati Uniti attraverso il SARS-CoV-2Sequenziamento per la risposta alle emergenze sanitarie pubbliche,Epidemiologia e sorveglianza (SPHERES)§§consorzio,che comprende circa 170 istituzioni partecipanti e promuove la condivisione aperta dei dati per facilitare l'uso di SARS-CoV-2dati di sequenzaPer tracciare l'evoluzione virale SARS-CoV-2, CDC èimplementare una sorveglianza genomica multiforme per capirei processi epidemiologici, immunologici ed evolutiviche danno forma alle filogenesi virali (filodinamica);focolaio guidaindagini;e facilitare la rilevazione e caratterizzazionezione di possibili reinfezioni, casi di innovazione vaccinale evarianti virali emergenti.Nel novembre 2020 è stato istituito CDCil programma nazionale di sorveglianza del ceppo SARS-CoV-2 (NS3).migliorare la rappresentatività della SARS-CoV-2 domesticasequenze.Il programma collabora con 64 pubblici statunitensilaboratori sanitari per supportare un sistema di sorveglianza genomica;NS3 sta anche costruendo una raccolta di campioni di SARS-CoV-2 and sequenze per supportare la risposta della sanità pubblica e scientificaricerca per valutare l'impatto delle relative mutazioni sucontromisure mediche raccomandate esistenti.CDC haha anche stipulato un contratto con diversi grandi laboratori clinici commercialitory per sequenziare rapidamente decine di migliaia di SARS-CoV-2–campioni positivi ogni mese e ha finanziato sette accademiciistituzioni per condurre la sorveglianza genomica in partenariatocon le agenzie di sanità pubblica, aggiungendo così sostanzialmente ala disponibilità di dati tempestivi di sorveglianza genomica provenienti da tutto il mondogli Stati Uniti.Oltre a queste iniziative nazionali,molte agenzie di sanità pubblica statali e locali stanno sequenziando
FIGURA 1. Traiettorie di incidenza del caso simulate* delle attuali varianti di SARS-CoV-2 e della variante B.1.1.7,†assumendo nessuna vaccinazione comunitariae iniziale R t = 1,1 (A) o iniziale R t = 0,9 (B) per le varianti correnti—Stati Uniti, gennaio–aprile 2021
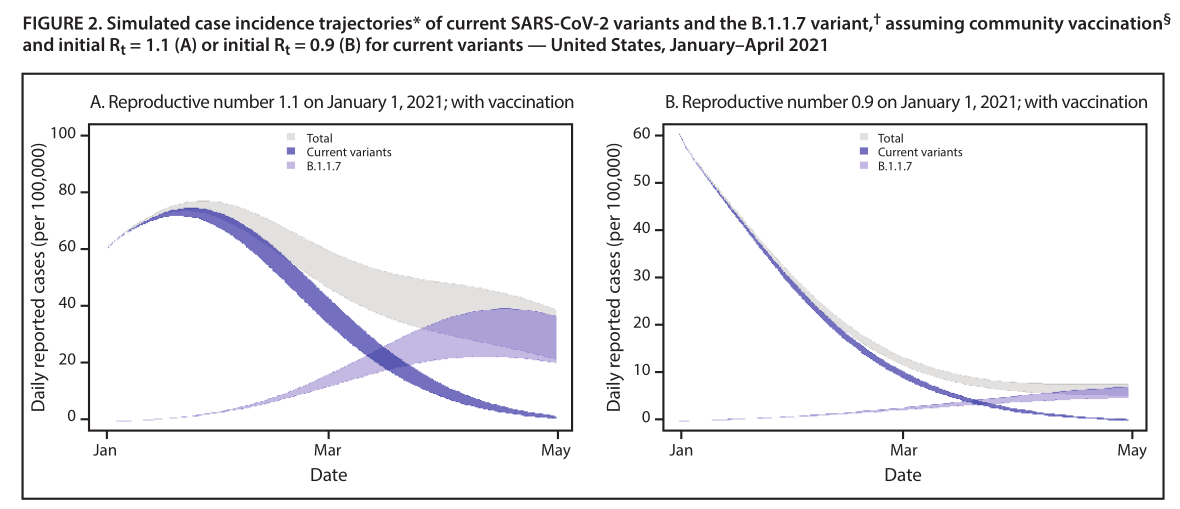


SARS-CoV-2 per comprendere meglio l'epidemiologia locale esostenere la risposta della sanità pubblica alla pandemia.I risultati di questa relazione sono soggetti ad almeno tre limitizioni.In primo luogo, l'entità dell'aumento della trasmissioneità negli Stati Uniti rispetto a quello osservato nelIl Regno Unito rimane poco chiaro.In secondo luogo, la prevalenza diB.1.1.7 negli Stati Uniti è anche sconosciuto in questo momento, mail rilevamento delle varianti e la stima della prevalenza migliorerannocon maggiori sforzi di sorveglianza degli Stati Uniti.Infine, mitigazione localeanche le misure di zione sono molto variabili, portando a variazioni inRt.I risultati specifici qui presentati si basano su simulazioni e non ha ipotizzato alcun cambiamento nelle mitigazioni oltre il 1° gennaio.La maggiore trasmissibilità della guerra variante B.1.1.7inveisce alla rigorosa attuazione delle strategie di salute pubblica perridurre la trasmissione e ridurre il potenziale impatto di B.1.1.7,guadagnare tempo critico per aumentare la copertura vaccinale.CDCi dati di modellazione mostrano che l'uso universale e l'aumento di compliance con misure di mitigazione e vaccinazione sono cruciali perridurre sostanzialmente il numero di nuovi casi e decessi nelprossimi mesi.Ulteriori test strategici di persone senzasintomi di COVID-19, ma che sono a maggior rischio diinfezione da SARS-CoV-2, offre un'altra opportunità perlimitare la diffusione in corso.Collettivamente, sorveglianza genomica potenziatalancia combinato con una maggiore conformità con la salute pubblicastrategie di mitigazione, tra cui vaccinazione, distanza fisicaing, uso di mascherine, igiene delle mani, isolamento e quarantena,sarà essenziale per limitare la diffusione di SARS-CoV-2 etutelare la salute pubblica.
Ringraziamenti
Membri del Sequenziamento per l'Emergenza Sanitaria PubblicaConsorzio di risposta, epidemiologia e sorveglianza;statale e localelaboratori di sanità pubblica;Associazione dei Laboratori di Sanità Pubblica;Squadra di risposta CDC COVID-19;Ramo dei virus respiratori,Divisione delle malattie virali, CDC.Committee of Medical Journal Editors modulo per la divulgazione del potenzialeconflitto di interessi.Nessun potenziali conflitti di interesse sono stati resi noti.
Riferimenti
1. Sanità pubblica Inghilterra.Investigation of novel SARS-CoV-2 variant: variant of concern 202012/01, briefing tecnico 3. Londra, Regno Unito: Public Health England;2020. https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/950823/Variant_of_Concern_VOC_202012_01_Technical_Briefing_3_-_England.pdf
2. Kemp SA, Harvey WT, Datir RP, et al.Emergenza e trasmissione ricorrenti di una delezione del picco SARS-CoV-2 ΔH69/V70.bioRxiv[Preprint pubblicato online il 14 gennaio 2021].https://www.biorxiv.org/content/10.1101/2020.12.14.422555v4
3. Volz E, Mishra S, Chand M, et al.Trasmissione del lignaggio SARS-CoV-2 B.1.1.7 in Inghilterra: approfondimenti dal collegamento di dati epidemiologici e genetici.medRxiv [Preprint pubblicato online il 4 gennaio 2021].https://www.medrxiv.org/content/10.1101/2020.12.30.20249034v2
4. Honein MA, Christie A, Rose DA, et al.;Squadra di risposta CDC COVID-19.Sintesi delle linee guida per le strategie di sanità pubblica per affrontare gli alti livelli di trasmissione comunitaria di SARS-CoV-2 e decessi correlati, dicembre 2020. MMWR Morb Mortal Wkly Rep 2020;69:1860–7.PMID:33301434 https://doi.org/10.15585/mmwr.mm6949e2
5. Volz E, Hill V, McCrone JT, et al.;Consorzio COG-UK.Valutazione degli effetti della mutazione della punta SARS-CoV-2 D614G su trasmissibilità e patogenicità.Cella 2021;184:64–75.e11.PMID:33275900 https://doi.org/10.1016/j.cell.2020.11.020
6. Korber B, Fischer WM, Gnanakaran S, et al.;Gruppo di genomica di Sheffield COVID-19.Monitoraggio dei cambiamenti nel picco SARS-CoV-2: prova che D614G aumenta l'infettività del virus COVID-19.Cellula
2020;182:812–27.PMID:32697968 https://doi.org/10.1016/j.cell.2020.06.043
7. McCarthy KR, Rennick LJ, Namnulli S, et al.Le delezioni naturali nella glicoproteina del picco SARS-CoV-2 guidano la fuga dell'anticorpo.bioRxiv [Preprint pubblicato online il 19 novembre 2020].https://www.biorxiv.org/content/
10.1101/2020.11.19.389916v18.I pattern di abbandono del gene Washington NL, White S, Schiabor KM, Cirulli ET, Bolze A, Lu JT.S nei test SARS-CoV-2 suggeriscono la diffusione della mutazione H69del/V70del negli Stati Uniti.medRxiv [Preprint pubblicato online il 30 dicembre 2020].https://www.medrxiv.org/content/10.1101/2020.12.24.20248814v1
9. Weisblum Y, Schmidt F, Zhang F, et al.Fuga dagli anticorpi neutralizzanti grazie alle varianti della proteina spike SARS-CoV-2.eLife 2020;9:e61312.PMID:33112236 https://doi.org/10.7554/eLife.61312
10. Greaney AJ, Loes AN, Crawford KHD, et al.Mappatura completa delle mutazioni nel dominio di legame del recettore SARS-CoV-2 che influenzano il riconoscimento da parte degli anticorpi policlonali del siero umano.bioRxiv [Preprint pubblicato online il 4 gennaio 2021].https://www.biorxiv.org/content/10.1101/2020.12.31.425021v1
Tempo di pubblicazione: febbraio-11-2021








