La tecnologia di diagnosi molecolare utilizza metodi di biologia molecolare per rilevare l'espressione e la struttura del materiale genetico del corpo umano e vari agenti patogeni, in modo da raggiungere lo scopo di prevedere e diagnosticare malattie.
Negli ultimi anni, con l'aggiornamento e l'iterazione della tecnologia diagnostica molecolare, l'applicazione clinica della diagnostica molecolare è diventata sempre più ampia e approfondita e il mercato della diagnostica molecolare è entrato in un periodo di rapido sviluppo.
L'autore riassume le tecnologie diagnostiche molecolari comuni sul mercato ed è diviso in tre parti: la prima parte introduce la tecnologia PCR, la seconda parte introduce la tecnologia di amplificazione isotermica dell'acido nucleico e la seconda parte introduce la tecnologia di sequenziamento.
01
Parte I: Tecnologia PCR
Tecnologia PCR
La PCR (reazione a catena della polimerasi) è una delle tecnologie di amplificazione del DNA in vitro, con una storia di oltre 30 anni.
La tecnologia PCR è stata sperimentata nel 1983 da Kary Mullis di Cetus, USA.Mullis fece domanda per un brevetto PCR nel 1985 e nello stesso anno pubblicò il primo documento accademico PCR sulla scienza.Mullis ha vinto il premio Nobel per la chimica nel 1993.
Principi di base della PCR
La PCR può amplificare i frammenti di DNA bersaglio di oltre un milione di volte.Il principio è che sotto la catalisi della DNA polimerasi, il DNA del filamento genitore viene utilizzato come modello e un primer specifico viene utilizzato come punto di partenza per l'estensione.Viene replicato in vitro attraverso passaggi come denaturazione, ricottura ed estensione.Il processo del DNA del filamento figlia complementare al DNA stampo del filamento genitore.
Il processo PCR standard è suddiviso in tre fasi:
1. Denaturazione: utilizzare l'alta temperatura per separare i doppi filamenti di DNA.I legami idrogeno tra i doppi filamenti del DNA si rompono ad alte temperature (93-98°C).
2. Ricottura: dopo che il DNA a doppio filamento è stato separato, la temperatura viene abbassata in modo che il primer possa legarsi al DNA a singolo filamento.
3. Estensione: la DNA polimerasi inizia a sintetizzare filamenti complementari lungo i filamenti di DNA dai primer legati quando la temperatura viene abbassata.Quando l'estensione è completata, un ciclo è completato e il numero di frammenti di DNA raddoppia.
Alternando questi tre passaggi 25-35 volte, il numero di frammenti di DNA aumenterà esponenzialmente.
L'ingegnosità della PCR è che diversi primer possono essere progettati per diversi geni bersaglio, in modo che i frammenti del gene bersaglio possano essere amplificati in un breve periodo di tempo.
Finora, la PCR può essere suddivisa in tre categorie, ovvero la PCR ordinaria, la PCR quantitativa fluorescente e la PCR digitale.
La prima generazione di PCR ordinaria
Utilizzare un normale strumento di amplificazione PCR per amplificare il gene bersaglio, quindi utilizzare l'elettroforesi su gel di agarosio per rilevare il prodotto, è possibile eseguire solo analisi qualitative.
I principali svantaggi della PCR di prima generazione:
-Soggetto ad amplificazione non specifica e risultati falsi positivi.
-Il rilevamento richiede molto tempo e l'operazione è macchinosa.
-Si possono eseguire solo test qualitativi.
PCR quantitativa in fluorescenza di seconda generazione
La PCR quantitativa a fluorescenza (Real-Time PCR), nota anche come qPCR, viene utilizzata per monitorare l'accumulo di prodotti amplificati attraverso l'accumulo di segnali fluorescenti aggiungendo sonde fluorescenti che possono indicare l'andamento del sistema di reazione e per giudicare i risultati attraverso la curva di fluorescenza e può essere quantificato con l'aiuto del valore Cq e della curva standard.
Poiché la tecnologia qPCR viene eseguita in un sistema chiuso, la probabilità di contaminazione è ridotta e il segnale di fluorescenza può essere monitorato per il rilevamento quantitativo, quindi è la più utilizzata nella pratica clinica ed è diventata la tecnologia dominante nella PCR.
Le sostanze fluorescenti utilizzate nella PCR quantitativa fluorescente in tempo reale possono essere suddivise in: sonde fluorescenti TaqMan, fari molecolari e coloranti fluorescenti.
1) Sonda fluorescente TaqMan:
Durante l'amplificazione PCR, viene aggiunta una sonda fluorescente specifica durante l'aggiunta di una coppia di primer.La sonda è un oligonucleotide e le due estremità sono rispettivamente marcate con un gruppo fluorescente reporter e un gruppo fluorescente quencher.
Quando la sonda è integra, il segnale fluorescente emesso dal gruppo reporter viene assorbito dal gruppo quenching;durante l'amplificazione della PCR, l'attività esonucleasica 5′-3′ dell'enzima Taq scinde e degrada la sonda, rendendo il reporter gruppo fluorescente e quencher Il gruppo fluorescente viene separato, in modo che il sistema di monitoraggio della fluorescenza possa ricevere il segnale di fluorescenza, cioè, ogni volta che un filamento di DNA viene amplificato, si forma una molecola fluorescente e l'accumulo del segnale di fluorescenza è completamente sincronizzato con la formazione del prodotto PCR.
2) Coloranti fluorescenti SYBR:
Nel sistema di reazione PCR viene aggiunto un eccesso di colorante fluorescente SYBR.Dopo che il colorante fluorescente SYBR è stato incorporato in modo non specifico nel doppio filamento di DNA, emette un segnale fluorescente.La molecola di colorante SYBR che non è incorporata nella catena non emetterà alcun segnale fluorescente, garantendo così il segnale fluorescente. L'aumento dei prodotti PCR è completamente sincronizzato con l'aumento dei prodotti PCR.SYBR si lega solo al DNA a doppio filamento, quindi la curva di fusione può essere utilizzata per determinare se la reazione PCR è specifica.
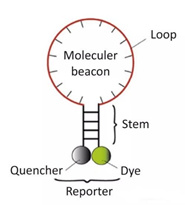
3) Segnali molecolari
È una sonda oligonucleotidica stem-loop a doppia marcatura che forma una struttura a forcina di circa 8 basi alle estremità 5 e 3.Le sequenze di acido nucleico ad entrambe le estremità sono accoppiate in modo complementare, causando la stretta chiusura del gruppo fluorescente e del gruppo di spegnimento.Chiuso, non produrrà fluorescenza.
Dopo la generazione del prodotto PCR, durante il processo di ricottura, la parte centrale del segnale molecolare viene accoppiata con una specifica sequenza di DNA e il gene fluorescente viene separato dal gene quencher per produrre fluorescenza.
I principali svantaggi della PCR di seconda generazione:
La sensibilità è ancora carente e il rilevamento di campioni a basso numero di copie non è accurato.
C'è un'influenza del valore di fondo e il risultato è suscettibile di interferenza.
PCR digitale di terza generazione
La PCR digitale (DigitalPCR, dPCR, Dig-PCR) calcola il numero di copie della sequenza target attraverso il rilevamento del punto finale e può eseguire un rilevamento quantitativo assoluto accurato senza utilizzare controlli interni e curve standard.
La PCR digitale utilizza il rilevamento dell'endpoint e non dipende dal valore Ct (soglia del ciclo), quindi la reazione PCR digitale è meno influenzata dall'efficienza di amplificazione e la tolleranza agli inibitori della reazione PCR è migliorata, con elevata precisione e riproducibilità.
A causa delle caratteristiche di alta sensibilità e alta precisione, non è facilmente interferito dagli inibitori della reazione PCR e può ottenere una vera quantificazione assoluta senza prodotti standard, che è diventato un punto di riferimento per la ricerca e l'applicazione.
Secondo le diverse forme dell'unità di reazione, può essere suddivisa in tre tipi: sistemi microfluidici, chip e goccioline.
Tempo di pubblicazione: luglio-08-2021