1. Rilevare l'assorbanza della soluzione di RNA
L'assorbanza a 280, 320, 230 e 260 nm rappresenta rispettivamente i valori di acido nucleico, fondo (torbidità della soluzione), concentrazione salina e materia organica come le proteine.Generalmente guarda solo OD260/OD280 (Rapporto, R).Quando 1,8 ~ 2,0, pensiamo che la contaminazione di proteine o altra materia organica nell'RNA possa essere tollerata, ma va notato che quando Tris viene utilizzato come tampone per rilevare l'assorbanza, il valore R può essere maggiore di 2 (generalmente dovrebbe essere <2,2).Quando R<1.8, l'inquinamento di proteine o altra materia organica nella soluzione è più evidente e il destino dell'RNA può essere determinato in base alle esigenze.Quando R>2.2, significa che l'RNA è stato idrolizzato in un singolo acido nucleico.
2. Schema elettroforetico dell'RNA
Generalmente, il gel denaturante viene utilizzato per l'elettroforesi dell'RNA, ma se è solo per rilevare la qualità dell'RNA, il gel denaturante non è necessario e può essere utilizzato il normale gel di agarosio.Lo scopo dell'elettroforesi è rilevare l'integrità delle bande 28S e 18S e il loro rapporto, o l'integrità dello striscio di mRNA.Generalmente, se le bande 28S e 18S sono luminose, chiare e nitide (riferendosi ai bordi delle bande sono chiare) e la luminosità di 28S è più del doppio di quella della banda 18S, consideriamo buona la qualità dell'RNA.
Quanto sopra sono i due metodi che usiamo comunemente, ma nessuno di questi due metodi può dirci chiaramente se c'è RNasi residua nella soluzione di RNA.Se c'è una quantità molto piccola di RNasi nella soluzione, è difficile per noi rilevarla con il metodo di cui sopra, ma la maggior parte delle successive reazioni enzimatiche vengono eseguite a temperature superiori a 37 gradi e per lungo tempo.In questo modo, se c'è una quantità molto piccola di RNasi nella soluzione di RNA, allora ci sarà un ambiente e un tempo molto adatti per svolgere il loro ruolo negli esperimenti successivi, e ovviamente l'esperimento sarà freddo in questo momento.Di seguito introduciamo un metodo che può confermare se vi è RNasi residua nella soluzione di RNA.
3. Test di conservazione del calore
In base alla concentrazione del campione, prelevare due RNA da 1000 ng dalla soluzione di RNA e aggiungerlo a una provetta da centrifuga da 0,5 ml e integrarlo con tampone Tris a pH 7,0 fino a un volume totale di 10 ul, quindi sigillare il tappo della provetta.Mettetene uno a bagnomaria a temperatura costante a 70°C e tenetelo al caldo per 1 h.L'altra parte è stata conservata in frigorifero a -20°C per 1 ora.Quando il tempo è scaduto, rimuovere i due campioni per l'elettroforesi.Al termine dell'elettroforesi, confrontare le bande elettroforetiche dei due.Se le bande dei due sono coerenti o non presentano differenze significative (ovviamente, anche le loro bande soddisfano le condizioni del metodo 2), significa che non vi è alcuna contaminazione residua da RNasi nella soluzione di RNA e la qualità dell'RNA è molto buona.Al contrario, se il campione incubato a 70°C mostra un'evidente degradazione, indica che c'è contaminazione da RNasi nella soluzione di RNA.
2 Metodi e tecniche sperimentali per l'estrazione dell'RNA
I problemi che incontriamo spesso durante l'estrazione dell'RNA sono: (1) la resa dell'RNA è bassa;(2) l'RNA ha un grave inquinamento salino;(3) l'RNA ha un grave inquinamento da solventi organici;(4) degradazione del campione e altri problemi
1. Reagenti di estrazione dell'RNA totale comunemente usati
Il metodo dell'isotiocianato di guanidina e il metodo Trizol sono i metodi più comunemente utilizzati per l'estrazione dell'RNA totale da tessuti e cellule animali.È particolarmente adatto per piccoli campioni e tessuti particolarmente difficili da estrarre, come l'estrazione di RNA totale da pelle di coniglio e tessuto connettivo animale;inoltre, Trizol, come reagente di lisi generico, può essere utilizzato anche per l'estrazione di tessuti vegetali, batteri, funghi e altri tessuti.Per i tessuti vegetali contenenti polisaccaridi e polifenoli, come camelia oleifera, foglie di tè, colza, ecc., il metodo CTAB può essere utilizzato anche per estrarre l'RNA totale.
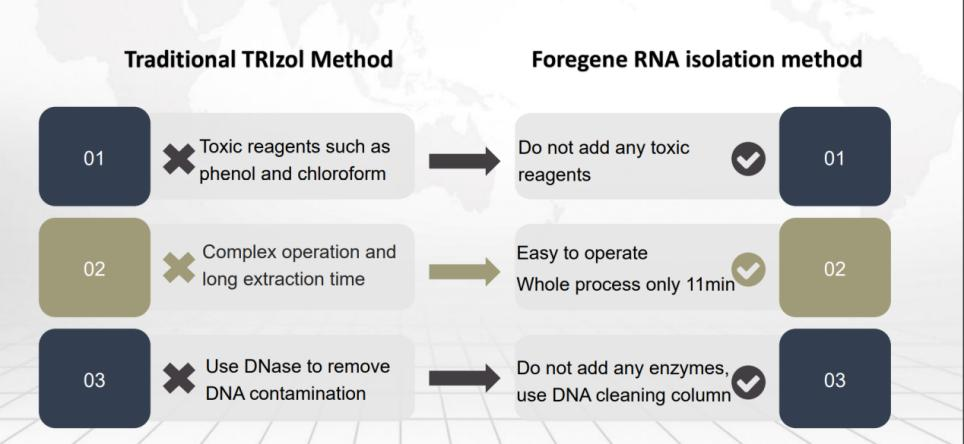
Come metodo convenzionale, anche il metodo a doppia colonna è molto popolare grazie al suo normale funzionamento a temperatura, all'assenza di necessità di aggiungere RNasi e alla sicurezza: assenza di cloroformio, fenoli e altri reagenti organici per l'estrazione.(prodotti consigliati )

2. Estrazione dell'RNA totale da tessuti animali
(1) Prova a scegliere tessuto fresco, se non è fresco (preferibilmente entro tre mesi - 80 ℃ frigorifero o congelato in azoto liquido. Quando tagli il tessuto, non tagliare direttamente a temperatura ambiente, assicurati di metterlo sulla ghiacciaia, cerca di evitare ripetuti congelamenti e scongelamenti.
(2) Utilizzare forbici e pinzette pulite per tagliare un piccolo pezzo di tessuto, provare a tagliare la parte centrale del tessuto durante il taglio del campione, oppure tagliare prima il pezzo di tessuto grande dal centro, quindi tagliare il campione nella nuova posizione di incisione.Il tessuto rimosso deve essere completamente sminuzzato, mettere il tessuto sminuzzato in un tubo EP senza RNase, aggiungere il lisato, il tessuto sminuzzato deve essere completamente esposto al lisato e prepararsi per l'omogeneizzazione.
(3) Per i tessuti normali, selezionare i tessuti delle dimensioni di un fagiolo mung (30-60 mg) per l'omogeneizzazione.Se i tessuti contengono una grande quantità di proteine, grassi o tessuti fibrosi densi come il fegato, aumentare o diminuire in modo appropriato la quantità di tessuti tagliati (facoltativo) Scegliere 10~20 mg).
(4) Se si estraggono muscolo di pesce, polpa di gamberetti, meduse e altri tessuti con un elevato contenuto di acqua, il volume del campione deve essere opportunamente aumentato (consigliato 100-200 mg).
(5) Se le condizioni lo consentono, il tessuto animale può essere estratto direttamente dopo essere stato omogeneizzato con un omogeneizzatore di tessuto ad alto passaggio, se non esiste tale attrezzatura.
(6) L'RNA ottenuto dopo l'estrazione finale deve essere posto immediatamente sulla ghiacciaia per ridurre la degradazione dell'RNA.
3. Estrazione dell'RNA da cellule animali
(1) Cellule in sospensione: centrifugare direttamente ed eliminare il terreno, lavare con PBS sterile per 1-2 volte, quindi sospendere con una quantità appropriata di PBS, quindi aggiungere il lisato per la lisi.Non aggiungere il lisato direttamente alle cellule precipitate dopo aver eliminato completamente il liquido.Ciò farà sì che il pacchetto di istoni rilasciato dopo che le cellule lisate sullo strato esterno aderiscano all'esterno delle cellule precipitate, limitando così il contatto delle cellule all'interno del pellet con il lisato., con conseguente lisi cellulare incompleta e ridotta resa di RNA.
(2) Cellule semi-aderenti o non strettamente aderenti: Dopo aver scartato il mezzo, lavare con PBS per 1-2 volte, quindi assorbire direttamente una quantità appropriata di PBS e soffiare il piatto di coltura con una pipetta o una pistola per eliminare le cellule e trasferirle in cellule prive di RNA.Aggiungere il lisato alla provetta EP dell'enzima per l'estrazione.
(3) Cellule aderenti: devono essere prima digerite con tripsina, quindi raccolte in provette EP prive di RNasi, centrifugate per rimuovere il surnatante, lavate 1-2 volte con PBS per rimuovere la tripsina in eccesso e risospese con una quantità appropriata di PBS Quindi procedere alla fase di estrazione.
4. Estrazione dell'RNA vegetale
I tessuti vegetali sono ricchi di composti fenolici, o ricchi di polisaccaridi, o contengono alcuni metaboliti secondari non identificati, o hanno un'elevata attività di RNasi.Queste sostanze sono strettamente combinate con l'RNA dopo la lisi cellulare per formare complessi insolubili o precipitati colloidali, che sono difficili da rimuovere.Pertanto, quando estraiamo il tessuto vegetale, dobbiamo scegliere un kit per piante.Il lisato nel kit può risolvere efficacemente i problemi di facile ossidazione dei polifenoli e separazione dei composti polisaccaridici e degli acidi nucleici.
(Per l'estrazione dell'RNA di piante di polifenoli polisaccaridi, prodotti consigliati:
(1) La buccia, la polpa, i semi, le foglie, ecc. della pianta devono essere completamente macinati in un mortaio.Durante il processo di macinazione, l'azoto liquido deve essere reintegrato in tempo per evitare la fusione del campione.Il campione macinato deve essere aggiunto rapidamente al lisato e agitato per evitare la degradazione dell'RNA.
(2) Per campioni ricchi di fibre come riso e foglie di grano, la quantità di estrazione dovrebbe essere opportunamente ridotta, altrimenti la macinazione e la lisi del tessuto non saranno complete, con conseguente bassa resa di RNA estratto.
(3) Per i tessuti vegetali con un elevato contenuto di acqua, come il frutto del melograno, il frutto dell'anguria, il frutto della pesca, ecc., la dimensione del campione dovrebbe essere opportunamente aumentata (100-200 mg è facoltativo).
(4) I tessuti vegetali, come foglie di piante, rizomi, frutti duri e altri materiali sono generalmente consigliati per utilizzare azoto liquido per maltare accuratamente gli ingredienti in un mortaio, quindi procedere alla fase di estrazione.Gli omogeneizzatori di tessuti convenzionali potrebbero non essere efficaci nell'omogeneizzare i tessuti vegetali e generalmente non sono raccomandati.
5. Precauzioni per l'estrazione dell'RNA
(1) I campioni di tessuto devono essere il più freschi possibile per evitare ripetuti congelamenti e scongelamenti.
(2) Il tessuto dovrebbe essere completamente macinato durante l'estrazione e la quantità di tessuto non dovrebbe essere troppo piccola, figuriamoci troppo.
(3) Dopo l'aggiunta del lisato deve essere concesso un tempo di incubazione sufficiente per lisare completamente il campione.
(4) Quando si utilizza il metodo Trizol per l'estrazione, il principio dell'assorbimento del supernatante dopo la stratificazione è "preferire inalare meno che inalare di più" e non deve estrarre fino allo strato intermedio, altrimenti causerà una grave contaminazione del DNA genomico.
(5) Durante il lavaggio, il liquido di lavaggio deve infiltrarsi completamente attorno alla parete del tubo per garantire un lavaggio accurato.
(6) Per il metodo di estrazione su colonna, oltre a staccare la colonna dopo il lavaggio, anche la colonna di adsorbimento deve essere collocata in un banco ultrapulito e soffiata per 5-10 minuti per far evaporare completamente il solvente organico a secchezza.
(7) All'ultima eluizione del metodo della colonna, dopo aver aggiunto l'acqua DEPC, deve essere incubato per 3-5 minuti, oppure l'acqua DEPC deve essere riscaldata in anticipo a 60°C per aumentare la resa dell'eluizione.Nel metodo tradizionale di scissione Trizol e precipitazione con isopropanolo, l'RNA finale viene dissolto in acqua DEPC, quindi dovrebbe essere dato un tempo appropriato per la dissoluzione e il fondo della provetta da centrifuga dovrebbe essere continuamente soffiato con una punta di pipetta.
3 three Cause e soluzioni per bassa concentrazione di RNA/scarsa qualità
1. Il rendimento è troppo basso
Il campione estratto è troppo basso, la quantità totale è insufficiente o il campione estratto è troppo e la lisi non è completa;il tessuto o le cellule di qualità adeguata devono essere utilizzati per l'estrazione, il pretrattamento del campione deve essere eseguito correttamente e la lisi deve essere sufficiente.
2. Residui del genoma
Quando si estrae con il metodo Trizol, quando il supernatante viene aspirato nello strato intermedio dopo la stratificazione, si verificherà una grave contaminazione del genoma;è necessario prestare particolare attenzione durante la stratificazione per evitare di risucchiare lo strato intermedio.Se per l'estrazione si utilizza il metodo della colonna, è possibile selezionare per l'estrazione un kit contenente DNasi I.L'acido nucleico adsorbito sulla membrana viene digerito direttamente con DNasi I, che può ridurre notevolmente i residui di DNA.
3. Degradazione dell'RNA
Può essere la degradazione del campione estratto stesso o la degradazione causata durante il processo di estrazione;per quanto possibile, dovrebbero essere utilizzati campioni freschi per l'estrazione dell'RNA e i campioni raccolti dovrebbero essere conservati in azoto liquido o in frigorifero a -80°C per tempo e dovrebbero essere evitati ripetuti congelamenti e scongelamenti.Nel processo di estrazione dell'RNA devono essere utilizzati puntali privi di RNasi/DNasi, provette da centrifuga e altri materiali.Il processo di estrazione dovrebbe essere il più veloce possibile.L'RNA estratto deve essere posto su una ghiacciaia e conservato a -80 nel tempo.Se l'RNA estratto deve essere rilevato mediante elettroforesi su gel, l'elettroforesi deve essere eseguita immediatamente dopo l'estrazione e il tampone dell'elettroforesi deve essere sostituito con uno appena preparato.
4. Residui di sale e solventi organici
I reagenti di estrazione contengono sali di fenolo e guanidina e la soluzione di lavaggio contiene etanolo.Durante il processo di estrazione, il lisato non è stato completamente assorbito e scartato e la soluzione di lavaggio non è stata completamente asciugata.I sali residui e i solventi organici sono dannosi per la successiva trascrizione inversa e PCR.Diversi gradi di inibizione, quindi il lisato di tessuto dovrebbe essere completamente rimosso durante il processo di estrazione e il lavaggio dovrebbe essere sufficiente in modo che le pareti circostanti del tubo possano essere lavate.Inoltre, il tubo viene svuotato e soffiato è un passaggio necessario, che ridurrà ulteriormente il residuo di materia organica.
Per ulteriori informazioni sull'estrazione dell'RNA, visitare il nostro sito Web:
www.foreivd.com per ulteriori informazioni.

Tempo di pubblicazione: dic-01-2022