Panoramica
Identificazione rapida delle piante transgeniche
Testo/Tong Yucheng
Operazione sperimentale/Han Ying
Redattore/Wen Youjun
Parole/1600+
Tempo di lettura suggerito/8-10 minuti
Identificazione rapida delle piante transgeniche
Come nuovo arrivato in laboratorio, non è un buon lavoro escludere le piante positive da un gruppo di piante con un basso tasso di conversione.In primo luogo, il DNA deve essere estratto da un gran numero di campioni uno per uno, quindi i geni estranei verranno rilevati mediante PCR.Tuttavia, i risultati sono spesso spazi vuoti e bande con alcuni elementi occasionalmente, ma è impossibile determinare se ci sono rilevamenti mancati o falsi rilevamenti..È molto impotente affrontare tale processo e risultati sperimentali?Non preoccuparti, il fratello ti insegna come eliminare le piante positive transgeniche in modo semplice e accurato.
Passo 1: Primer di rilevamento del design
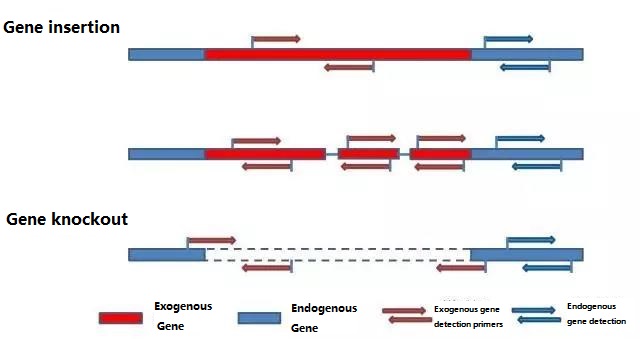
Determinare il gene endogeno e il gene esogeno da rilevare in base al campione da testare e selezionare una sequenza rappresentativa di 100-500 bp nel gene per la progettazione del primer.Buoni primer possono garantire l'accuratezza dei risultati di rilevamento e abbreviare il tempo di rilevamento (vedere l'appendice per i primer di rilevamento comunemente utilizzati).
Nota:
I primer di nuova concezione devono ottimizzare le condizioni di reazione e verificare l'accuratezza, la precisione e il limite di rilevamento del rilevamento prima di eseguire il rilevamento su larga scala.
Passo 2:Sviluppare il protocollo sperimentale

Controllo positivo: utilizzare il DNA purificato contenente il frammento bersaglio come modello per determinare se il sistema di reazione PCR e le condizioni sono normali.
Controllo negativo/bianco: utilizzare un templato di DNA o ddH2O che non contiene il frammento bersaglio come modello per rilevare se esiste una fonte di contaminazione nel sistema PCR.
Controllo di riferimento interno: utilizzare la combinazione primer/sonda del gene endogeno del campione da testare per valutare se il templato può essere rilevato mediante PCR.
Nota:
I controlli positivi, negativi/bianchi e i controlli di controllo interno devono essere impostati per ciascun test per valutare la validità dei risultati sperimentali.
Passaggio 3: Preparazione dell'esperimento

Prima dell'uso, osservare se la soluzione è uniformemente miscelata.Se viene rilevata la precipitazione, deve essere sciolta e miscelata secondo le istruzioni prima dell'uso.La miscela 2×PCR deve essere pipettata e miscelata ripetutamente con una micropipetta prima dell'uso per evitare una distribuzione irregolare degli ioni.
Nota:
Estrarre le istruzioni e leggerle attentamente e fare i preparativi prima dell'esperimento in stretta conformità con le istruzioni.
Passaggio 4: preparare il sistema di reazione PCR
Secondo il protocollo sperimentale, mescolare i primer, H2O, 2×PCR mix, centrifugare e distribuirli in ciascuna provetta di reazione.
Nota:
Per test su larga scala oa lungo termine, si consiglia di utilizzare un sistema di reazione PCR contenente l'enzima UNG, che può evitare efficacemente la contaminazione da aerosol causata dai prodotti PCR.
Passaggio 5: aggiungere il modello di reazione

Utilizzando la tecnologia Direct PCR, non è necessario il noioso processo di purificazione dell'acido nucleico.Il modello campione può essere preparato entro 10 minuti e aggiunto al corrispondente sistema di reazione PCR.
Nota:
Il metodo Lysis ha un migliore effetto di rilevamento e il prodotto ottenuto può essere utilizzato per più reazioni di rilevamento.
5.1: PCR diretta delle foglie
In base alle dimensioni dell'immagine nel manuale, tagliare il tessuto fogliare con un diametro di 2-3 mm e posizionarlo nel sistema di reazione PCR.
Nota: assicurarsi che i frammenti fogliari siano completamente immersi nella soluzione di reazione PCR e non aggiungere tessuto fogliare eccessivo.
5.2: Metodo della lisi fogliare
Tagliare il tessuto fogliare con un diametro di 5-7 mm e metterlo in una provetta da centrifuga.Se scegli foglie mature, evita di utilizzare i tessuti della nervatura principale della foglia.Pipettare 50 ul di tampone P1 lisato in una provetta da centrifuga per assicurarsi che il lisato possa immergere completamente il tessuto fogliare, collocarlo in un termociclatore o in un bagno di metallo e lisare a 95°C per 5-10 minuti.


Aggiungere 50ul di soluzione di neutralizzazione Buffer P2 e mescolare bene.Il lisato risultante può essere utilizzato come modello e aggiunto al sistema di reazione PCR.
Nota: la quantità di templato deve essere compresa tra il 5 e il 10% del sistema PCR e non deve superare il 20% (ad esempio, in un sistema PCR da 20 μl, aggiungere 1-2 μl di tampone di lisi, non più di 4 μl).
Passaggio 6: reazione PCR

Dopo aver centrifugato la provetta di reazione per PCR, inserirli in uno strumento per PCR per l'amplificazione.
Nota:
La reazione utilizza un modello non purificato per l'amplificazione, quindi il numero di cicli di amplificazione è di 5-10 cicli in più rispetto a quando si utilizza un modello di DNA purificato.
Passaggio 7: rilevamento dell'elettroforesi e analisi dei risultati

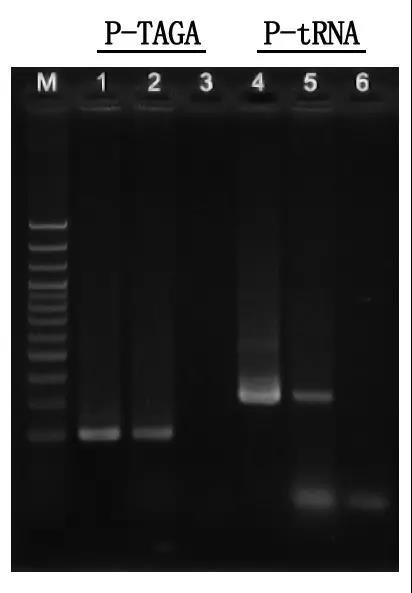
M: scala del DNA da 100 bp
1\4: Metodo del DNA purificato
2\5: metodo PCR diretto
3\6: Controllo vuoto
Controllo di qualità:
I risultati del test dei vari controlli impostati nell'esperimento devono soddisfare le seguenti condizioni.In caso contrario, la causa del problema dovrebbe essere analizzata e il test dovrebbe essere eseguito nuovamente dopo che il problema è stato eliminato.
Tabella 1. Risultati dei test normali di vari gruppi di controllo

*Quando il plasmide viene utilizzato come controllo positivo, il risultato del test del gene endogeno può essere negativo
Giudizio del risultato:
R. Il risultato del test del gene endogeno del campione è negativo, indicando che il DNA adatto per il normale rilevamento PCR non può essere estratto dal campione o che il DNA estratto contiene inibitori della reazione PCR e che il DNA deve essere estratto nuovamente.
B. Il risultato del test del gene endogeno del campione è positivo e il risultato del test del gene esogeno è negativo, indicando che il DNA adatto per il normale rilevamento della PCR viene estratto dal campione e si può ritenere che il gene XXX non viene rilevato nel campione.
C. Il risultato del test del gene endogeno del campione è positivo e il risultato del test del gene esogeno è positivo, indicando che il DNA adatto per il normale rilevamento della PCR è stato estratto dal campione e che il DNA del campione contiene il gene XXX.Gli esperimenti di conferma possono essere ulteriormente eseguiti.
Passaggio 8: progettare i primer di rilevamento

Dopo l'esperimento, utilizzare una soluzione di ipoclorito di sodio al 2% e una soluzione di etanolo al 70% per pulire l'area sperimentale per prevenire l'inquinamento ambientale.
Appendice
Tabella 2. Primer comunemente usati per il rilevamento PCR generale di piante geneticamente modificate
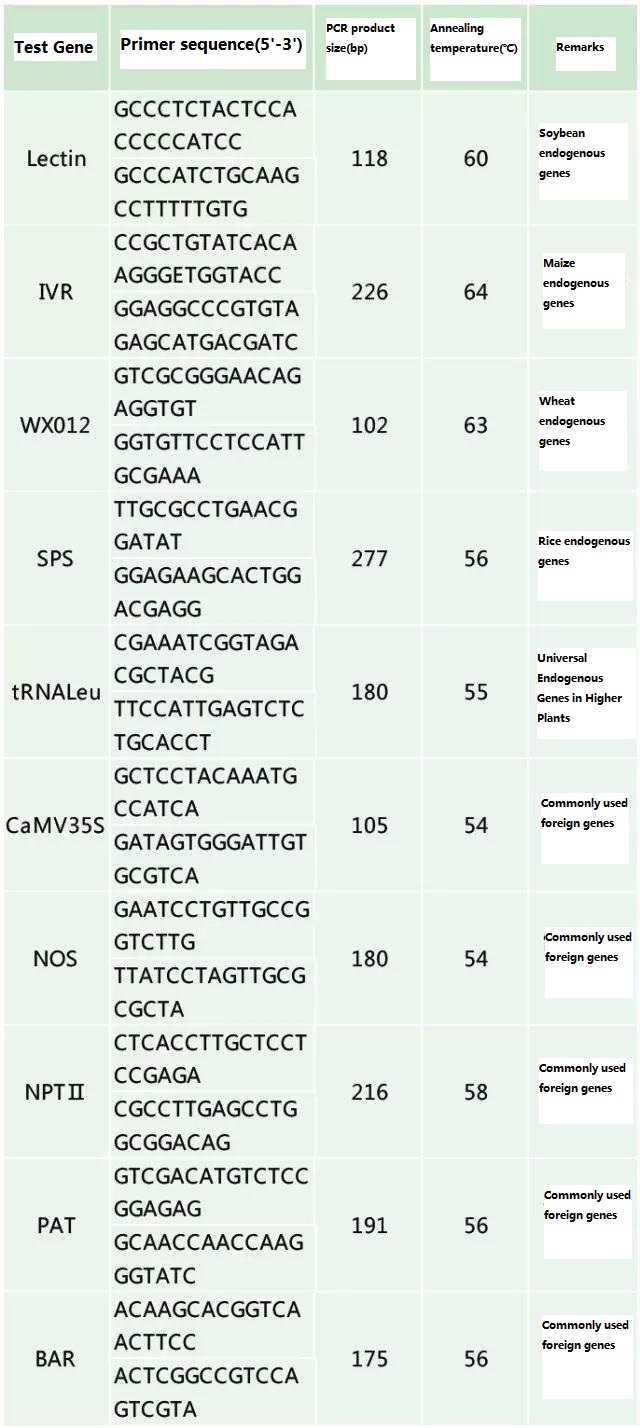
Documento di riferimento:
SN/T 1202-2010, metodo di rilevazione PCR qualitativa per ingredienti vegetali geneticamente modificati negli alimenti.
Ministero delle Politiche Agricole Bando 1485-5-2010, Sperimentazione degli ingredienti delle piante geneticamente modificate e dei loro prodotti-Riso M12 e suoi derivati.
Tempo di pubblicazione: giu-09-2021








