Materiale di partenza: RNA
La PCR di trascrizione inversa quantitativa (RT-qPCR) è un metodo sperimentale utilizzato negli esperimenti di PCR che utilizzano l'RNA come materiale di partenza.In questo metodo, l'RNA totale o RNA messaggero (mRNA) viene prima trascritto in DNA complementare (cDNA) mediante la trascrittasi inversa.Successivamente, è stata eseguita una reazione qPCR utilizzando il cDNA come modello.RT-qPCR è stato utilizzato in una varietà di applicazioni di biologia molecolare, tra cui analisi dell'espressione genica, convalida dell'interferenza dell'RNA, convalida di microarray, rilevamento di agenti patogeni, test genetici e ricerca sulle malattie.
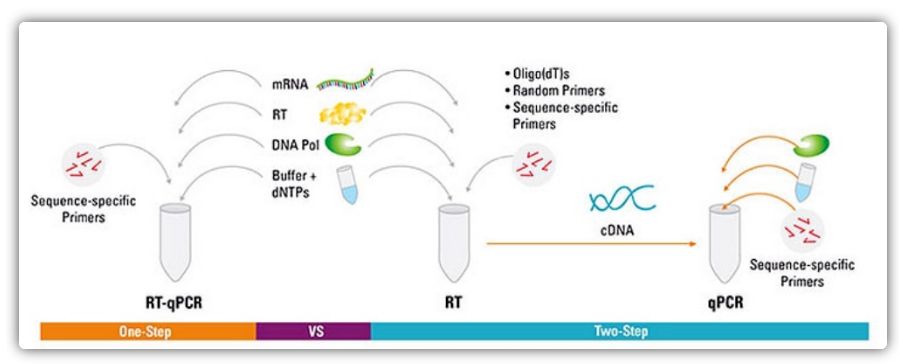
Metodi a una e due fasi per RT-qPCR
RT-qPCR può essere realizzato con un metodo a una o due fasi.La RT-qPCR in una fase combina la trascrizione inversa e l'amplificazione PCR, consentendo alla trascrittasi inversa e alla DNA polimerasi di completare la reazione nella stessa provetta nelle stesse condizioni tampone.One-step RT-qPCR richiede solo l'uso di primer specifici per sequenza.Nella RT-qPCR in due fasi, la trascrizione inversa e l'amplificazione PCR vengono eseguite in due provette, utilizzando diversi tamponi ottimizzati, condizioni di reazione e strategie di progettazione dei primer.
| Vantaggio | Svantaggio | |
| Un passo | Questo metodo ha meno errori sperimentali poiché entrambe le reazioni vengono eseguite in un tubo
Meno passaggi di pipettaggio riducono il rischio di contaminazione
Adatto per amplificazione/screening ad alto rendimento, veloce e riproducibile | Le reazioni in due fasi non possono essere ottimizzate separatamente
Poiché le condizioni di reazione sono compromesse combinando la reazione in due fasi, la sensibilità non è buona come quella del metodo in due fasi
Il numero di bersagli rilevati da un singolo campione è piccolo |
| Due Passi | Capacità di creare librerie di cDNA stabili che possono essere conservate per lunghi periodi di tempo e utilizzate in reazioni multiple
I geni bersaglio e i geni di riferimento possono essere amplificati dalla stessa libreria di cDNA senza la necessità di più librerie di cDNA
Tamponi di reazione e condizioni di reazione che consentono l'ottimizzazione di singole corse di reazione
Selezione flessibile delle condizioni di trigger | L'uso di più provette e più passaggi di pipettaggio aumenta il rischio di contaminazione del DNA, e richiede tempo.
Richiede una maggiore ottimizzazione rispetto al metodo in un solo passaggio |
Prodotti correlati:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
RT-qPCR Easyᵀᴹ (One Step)-Taqman
RT Easyᵀᴹ I Master Premix per la sintesi di CDNA di primo filamento
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Selezione di RNA totale e mRNA
Quando si progetta un esperimento RT-qPCR, è importante decidere se utilizzare l'RNA totale o l'mRNA purificato come modello per la trascrizione inversa.Sebbene l'mRNA possa essere in grado di fornire una sensibilità leggermente superiore, l'RNA totale è ancora frequentemente utilizzato.La ragione di ciò è che l'RNA totale ha un vantaggio più importante come materiale di partenza rispetto all'mRNA.In primo luogo, il processo richiede meno passaggi di purificazione, il che garantisce un migliore recupero quantitativo del modello e una migliore normalizzazione dei risultati rispetto ai numeri di cellule iniziali.In secondo luogo, evita la fase di arricchimento dell'mRNA, che può evitare la possibilità di risultati distorti a causa di diversi recuperi di diversi mRNA.Nel complesso, poiché nella maggior parte delle applicazioni la quantificazione relativa del gene bersaglio è più importante della sensibilità assoluta del rilevamento, l'RNA totale è più adatto nella maggior parte dei casi.
Primer di trascrizione inversa
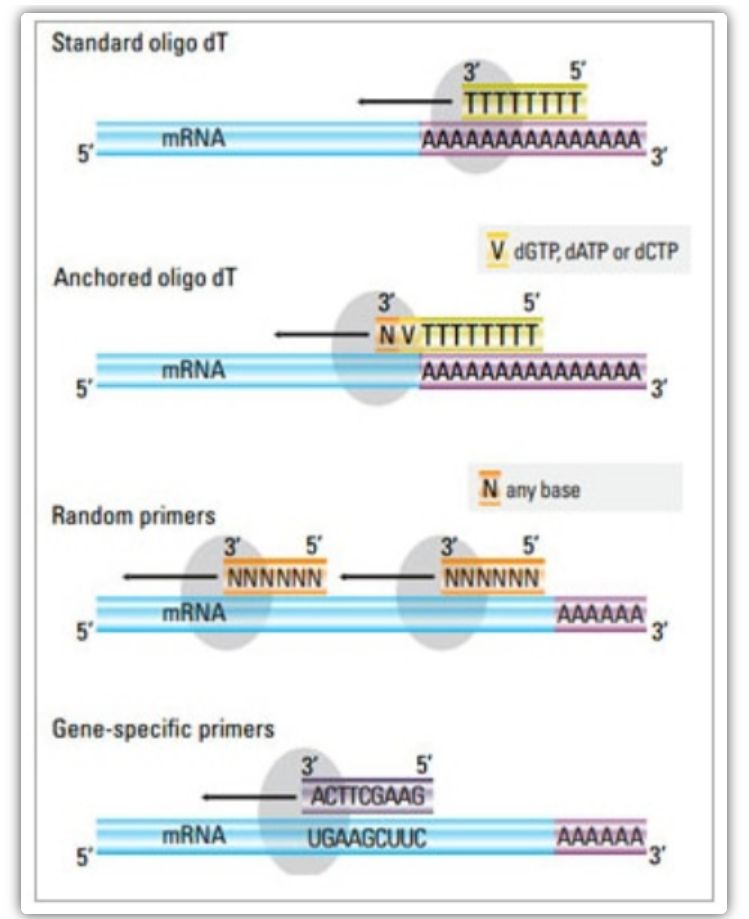
Nel metodo in due fasi, è possibile utilizzare tre diversi metodi per innescare la reazione del cDNA: primer oligo(dT), primer casuali o primer specifici per sequenza.Tipicamente, primer oligo(dT) e primer casuali sono usati in combinazione.Questi primer si appaiano al filamento di mRNA modello e forniscono alla trascrittasi inversa un punto di partenza per la sintesi.
| Selezione dell'innesco | Struttura e funzione | Vantaggio | Svantaggio |
| Primer oligo(dT) (o primer oligo(dT) ancorato) | Ricottura estesa ai residui di timina alla coda di poli (A) dell'mRNA;Il primer oligo(dT) di ancoraggio contiene un G, C o A all'estremità 3′ (sito di ancoraggio) | Sintesi di cDNA a lunghezza intera da mRNA a coda di poli (A).
Applicabile quando è disponibile meno materiale di partenza
Il sito di ancoraggio assicura che il primer oligo(dT) si leghi alla coda 5′ poli(A) dell'mRNA | Adatto solo per l'amplificazione di geni con code di poli(A).
Ottenere cDNA troncato dal sito di priming*2 in poli(A)
Prevenuto per legarsi all'estremità 3′*
*Questa possibilità è ridotta al minimo se si utilizzano primer oligo(dT) ancorati |
| primer casuale
| Da 6 a 9 basi di lunghezza, che possono associarsi a più siti durante la trascrizione dell'RNA | Anneal a tutti gli RNA (tRNA, rRNA e mRNA)
Adatto per trascrizioni con struttura secondaria significativa o quando è disponibile meno materiale di partenza
Alta resa cDNA | Il cDNA viene retrotrascritto da tutto l'RNA, che di solito non è desiderato e può diluire il segnale dell'mRNA bersaglio
ottenere cDNA troncato |
| primer sequenza-specifici | Primer personalizzati mirati a specifiche sequenze di mRNA | specifica libreria di cDNA
Migliora la sensibilità
Utilizzo di primer qPCR inverso | Limitato solo alla sintesi di un singolo gene bersaglio |
Trascrittasi inversa
La trascrittasi inversa è un enzima che utilizza l'RNA per sintetizzare il DNA.Alcune trascrittasi inverse hanno attività RNasi e possono degradare i filamenti di RNA nei filamenti ibridi RNA-DNA dopo la trascrizione.Se non ha attività enzimatica RNase, RNaseH può essere aggiunto per una maggiore efficienza qPCR.Gli enzimi comunemente usati includono la trascrittasi inversa del virus della leucemia murina Moloney e la trascrittasi inversa del virus del mieloblastoma aviario.Per RT-qPCR, è ideale scegliere una trascrittasi inversa con maggiore termostabilità, in modo che la sintesi del cDNA possa essere eseguita a temperature più elevate, garantendo la trascrizione di successo di RNA con struttura secondaria più elevata, pur mantenendo la loro piena attività durante la reazione, con conseguente maggiore resa di cDNA.
Prodotti correlati:
Foreasy M-MLV trascrittasi inversa
Attività RNasi H della trascrittasi inversa
RNaseH è in grado di degradare i filamenti di RNA dai duplex RNA-DNA, consentendo un'efficiente sintesi di DNA a doppio filamento.Tuttavia, quando si utilizza mRNA lungo come modello, l'RNA può essere degradato prematuramente, risultando in cDNA troncato.Pertanto, è spesso utile ridurre al minimo l'attività di RNaseH durante la clonazione del cDNA se si desidera la sintesi di trascritti lunghi.Al contrario, le trascrittasi inverse con attività RNase H sono spesso utili per le applicazioni qPCR perché migliorano la fusione dei duplex RNA-DNA durante il primo ciclo di PCR.
Disegno dell'innesco
I primer PCR utilizzati per la fase qPCR in RT-qPCR dovrebbero idealmente essere progettati per coprire una giunzione esone-esone, dove un primer di amplificazione potrebbe potenzialmente coprire un confine effettivo esone-introne.Poiché le sequenze di DNA genomico contenenti introni non sono amplificate, questo design riduce il rischio di falsi positivi amplificati dalla contaminazione del DNA genomico.
Se i primer non possono essere progettati per separare gli esoni o i confini esone-esone, potrebbe essere necessario trattare i campioni di RNA con DNasi I o dsDNasi senza RNasi per rimuovere la contaminazione del DNA genomico.
Controllo RT-qPCR
Un controllo negativo di trascrizione inversa (controllo -RT) dovrebbe essere incluso in tutti gli esperimenti RT-qPCR per rilevare la contaminazione del DNA (come DNA genomico o prodotti PCR da reazioni precedenti).Questo controllo contiene tutti i componenti della reazione tranne la trascrittasi inversa.Poiché la trascrizione inversa non si verifica con questo controllo, se si osserva l'amplificazione mediante PCR, è molto probabile la contaminazione da DNA.
Orario di pubblicazione: 02 agosto 2022








