Il vaccino a mRNA di Pfizer per COVID ha riacceso la passione per l'utilizzo dell'acido ribonucleico (RNA) come bersaglio terapeutico.Tuttavia, il targeting dell'RNA con piccole molecole è estremamente impegnativo.
L'RNA ha solo quattro elementi costitutivi: adenina (A), citosina (C), guanina (G) e uracile (U) che sostituisce la timina (T) presente nel DNA.Ciò rende la selettività dei farmaci un ostacolo quasi insormontabile.Al contrario, ci sono 22 aminoacidi naturali che costituiscono le proteine, il che spiega perché la maggior parte dei farmaci mirati alle proteine ha una selettività relativamente buona.
Struttura e funzione dell'RNA
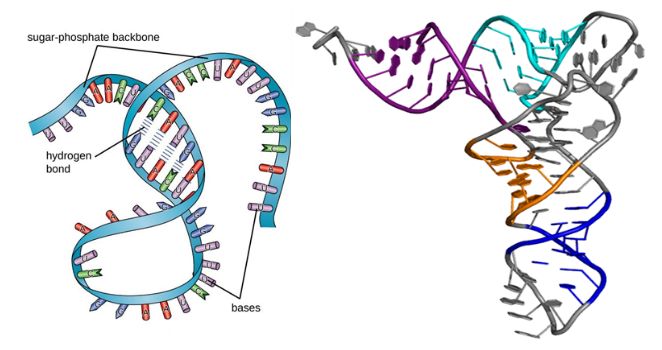
Come le proteine, le molecole di RNA hanno strutture secondarie e terziarie, come mostrato nella figura sottostante.Sebbene siano macromolecole a catena singola, la loro struttura secondaria prende forma quando l'accoppiamento delle basi provoca rigonfiamenti, anse ed eliche.Quindi, il ripiegamento tridimensionale porta alla struttura terziaria dell'RNA, che è essenziale per la sua stabilità e funzione.
Figura 1. Struttura dell'RNA
Esistono tre tipi di RNA:
- RNA messaggero (mRNA)trascrive l'informazione genetica dal DNA e viene trasferita come sequenza di basi sul ribosoma;l
- RNA ribosomiale (rRNA)fa parte degli organelli sintetizzatori di proteine chiamati ribosomi, che vengono esportati nel citoplasma e aiutano a tradurre le informazioni nell'mRNA in proteine;
- Trasferimento dell'RNA (tRNA)è il collegamento tra l'mRNA e la catena di amminoacidi che costituisce la proteina.
Mirare all'RNA come bersaglio terapeutico è molto attraente.È stato scoperto che solo l'1,5% del nostro genoma viene infine tradotto in proteine, mentre il 70%-90% viene trascritto in RNA.Le molecole di RNA sono le più importanti per tutti gli organismi viventi.Secondo il “dogma centrale” di Francis Crick, il ruolo più critico dell'RNA è quello di tradurre l'informazione genetica dal DNA in proteine.Inoltre, le molecole di RNA hanno anche altre funzioni, tra cui:
- Agendo come molecole adattatrici nella sintesi proteica;l
- Servendo da messaggero tra il DNA e il ribosoma;l
- Sono portatori di informazioni genetiche in tutte le cellule viventi;l
- Promuovere la selezione ribosomiale degli amminoacidi corretti, necessari per sintetizzare nuove proteinein vivo.
Antibiotici
Nonostante sia stato scoperto già negli anni '40, il meccanismo d'azione di molti antibiotici non è stato chiarito fino alla fine degli anni '80.Si è scoperto che un'ampia percentuale di antibiotici agisce legandosi ai ribosomi batterici per impedire loro di produrre proteine appropriate, uccidendo così i batteri.
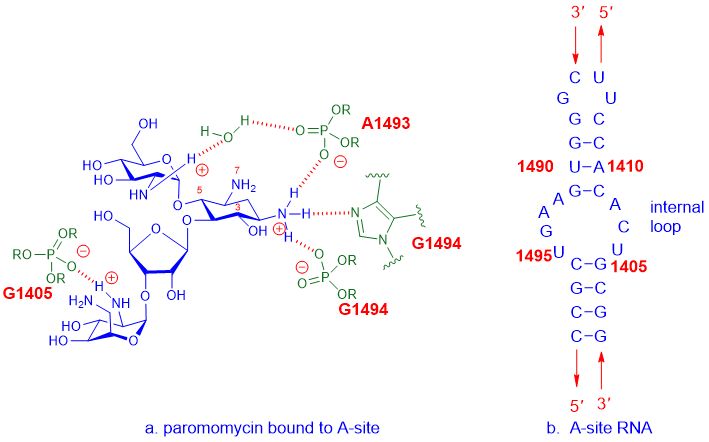
Ad esempio, gli antibiotici aminoglicosidici si legano al sito A dell'rRNA 16S, che fa parte della subunità ribosomiale 30S, e quindi interferiscono con la sintesi proteica per interferire con la crescita batterica, portando infine alla morte cellulare.Il sito A si riferisce al sito amminoacilico, noto anche come sito accettore di tRNA.L'interazione dettagliata tra farmaci aminoglicosidici, comeparomomicinae il sito A diEscherichia coliL'RNA è mostrato sotto.
Figura 2. L'interazione tra paromomicina e il sito A diEscherichia coliRNA
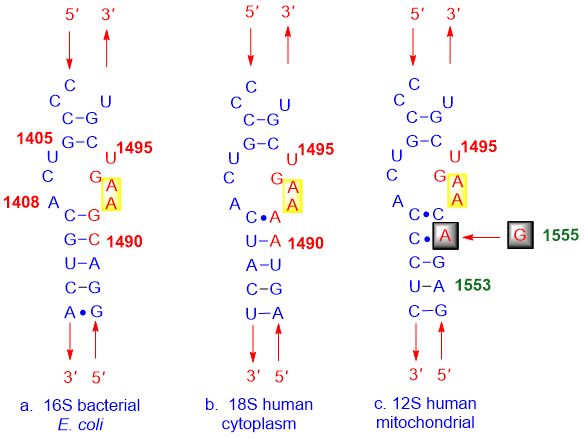
Sfortunatamente, molti inibitori del sito A, inclusi i farmaci aminoglicosidici, presentano problemi di sicurezza come nefrotossicità, ototossicità dose-dipendente e specifica irreversibile.Queste tossicità sono il risultato di una mancanza di selettività nei farmaci aminoglicosidici per il riconoscimento di piccole molecole di RNA.
Come mostrato nella figura seguente: (a) la struttura dei batteri, (b) la membrana cellulare umana e (c) il sito A mitocondriale umano sono molto simili, facendo sì che gli inibitori del sito A si leghino a tutti loro.
Figura 3. Il legame dell'inibitore del sito A non selettivo
Gli antibiotici tetracicline inibiscono anche il sito A dell'rRNA.Inibiscono selettivamente la sintesi proteica batterica legandosi in modo reversibile a una regione elicoidale (H34) sulla subunità 30S complessata con Mg2+.
D'altra parte, gli antibiotici macrolidi si legano vicino al sito di uscita (sito E) del tunnel ribosomiale batterico per i peptidi nascenti (NPET) e lo bloccano parzialmente, inibendo così la sintesi proteica batterica.Infine, gli antibiotici ossazolidinone comelinezolid(Zyvox) si legano a una profonda fenditura nella subunità ribosomiale batterica 50S, che è circondata da nucleotidi di rRNA 23S.
Oligonucleotidi antisenso (ASO)
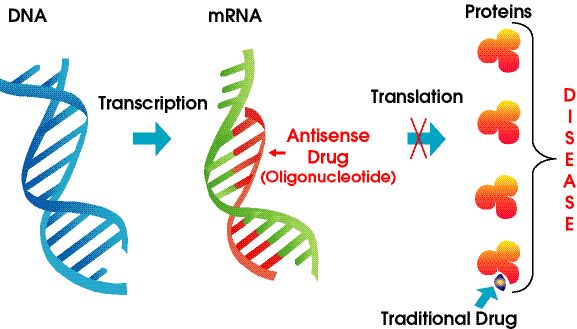
I farmaci antisenso sono polimeri di acidi nucleici modificati chimicamente che prendono di mira l'RNA.Si basano sull'accoppiamento di basi Watson-Crick per legarsi all'mRNA bersaglio, con conseguente silenziamento genico, blocco sterico o alterazione dello splicing.Gli ASO possono interagire con i pre-RNA nel nucleo cellulare e gli mRNA maturi nel citoplasma.Possono prendere di mira esoni, introni e regioni non tradotte (UTR).Ad oggi, più di una dozzina di farmaci ASO sono stati approvati dalla FDA.
Figura 4. Tecnologia antisenso
Farmaci a piccola molecola che prendono di mira l'RNA
Nel 2015, Novartis ha riferito di aver scoperto un regolatore di splicing SMN2 chiamato Branaplam, che migliora l'associazione di U1-pre-mRNA e salva i topi SMA.
Risdiplam (Evrysdi) di PTC/Roche è stato invece approvato dalla FDA nel 2020 per il trattamento della SMA.Come Branaplam, anche Risdiplam agisce regolando lo splicing dei geni SMN2 rilevanti per produrre proteine SMN funzionali.
Degradatori dell'RNA
RBM sta per proteina del motivo legante l'RNA.Essenzialmente, l'indolo sulfamidico è un adesivo molecolare.Recluta selettivamente RBM39 nell'ubiquitin ligasi CRL4-DCAF15 E3, promuovendo la poliubiquitinazione di RBM39 e la degradazione delle proteine.L'esaurimento genetico o la degradazione mediata da sulfamidici di RBM39 induce significative anomalie di splicing a livello di genoma, che alla fine portano alla morte cellulare.
Gli RNA-PROTAC sono sviluppati per degradare le proteine leganti l'RNA (RBP).PROTAC utilizza un linker per collegare il ligando della ligasi E3 al ligando dell'RNA, che si lega all'RNA e agli RBP.Poiché RBP contiene domini strutturali che possono legarsi a specifiche sequenze oligonucleotidiche, RNA-PROTAC utilizza una sequenza oligonucleotidica come ligando per la proteina di interesse (POI).Il risultato finale è il degrado degli RBP.
Recentemente, il professor Matthew Disney della Scripps Institution of Oceanography ha inventato l'RNAchimere mirate alla ribonucleasi (RiboTAC).RiboTAC è una molecola eterofunzionale che collega un ligando RNase L e un ligando RNA con un linker.Può reclutare in modo specifico l'RNasi L endogena verso specifici bersagli di RNA e quindi eliminare con successo l'RNA utilizzando il meccanismo di rottura dell'acido nucleico cellulare (RNasi L).
Man mano che i ricercatori apprendono di più sull'interazione tra piccole molecole e bersagli dell'RNA, in futuro emergeranno altri farmaci che utilizzano questo metodo.
Orario di pubblicazione: 02 agosto 2023