Il valore Ct è la forma di presentazione dei risultati più importante della PCR quantitativa fluorescente.Viene utilizzato per calcolare le differenze di espressione genica o il numero di copie geniche.Quindi qual è il valore Ct della quantificazione della fluorescenza considerato ragionevole?Come garantire l'intervallo effettivo del valore Ct?
Cos'è il valore Ct?
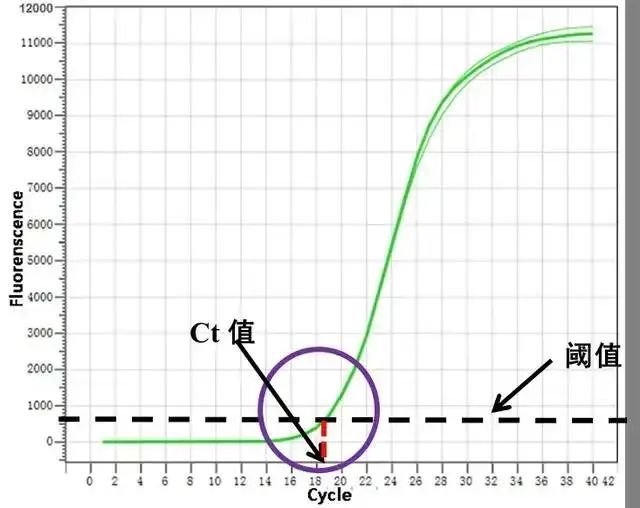
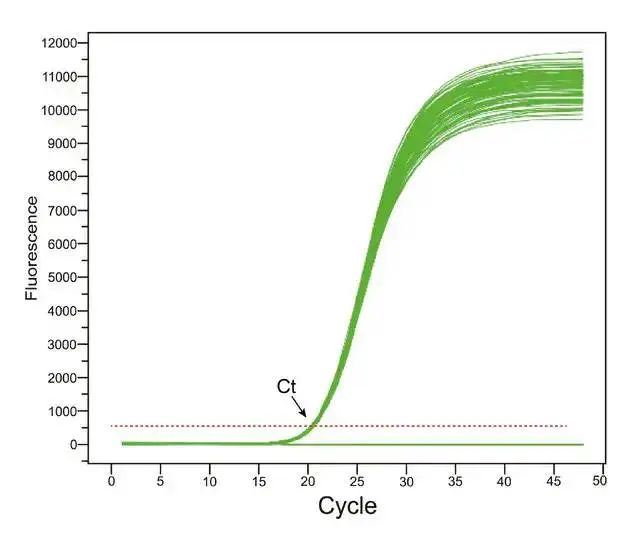
Durante il processo di amplificazione qPCR, il numero corrispondente di cicli di amplificazione (Cycle Threshold) quando il segnale di fluorescenza del prodotto amplificato raggiunge la soglia di fluorescenza impostata.C sta per Ciclo e T sta per Soglia.In poche parole, il valore Ct è il numero di cicli corrispondenti a quando l'amplificazione del modello iniziale raggiunge una certa quantità di prodotto in qPCR.La cosiddetta “una certa quantità di prodotto” sarà ulteriormente spiegata in seguito.
Cosa fa il valore Ct?
1. Relazione tra amplificazione esponenziale, quantità di templato e valore Ct
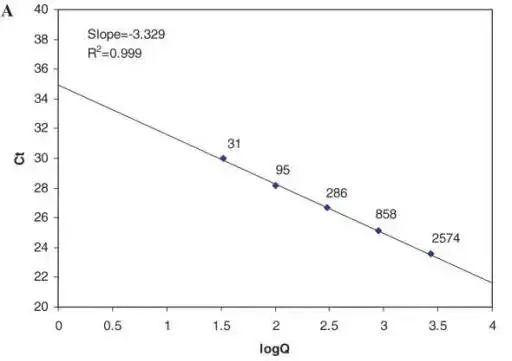
Idealmente, i geni in qPCR vengono accumulati dall'amplificazione esponenziale dopo un certo numero di cicli.La relazione tra il numero di cicli di amplificazione e la quantità di prodotti è: Quantità di prodotto amplificato = quantità iniziale del templato × (1+En) numero di cicli.Tuttavia, la reazione qPCR non è sempre in una situazione ideale.Quando la quantità di prodotto amplificato raggiunge una "determinata quantità di prodotto", il numero di cicli in questo momento è il valore Ct ed è nel periodo di amplificazione esponenziale.La relazione tra il valore Ct e la quantità del modello iniziale: esiste una relazione lineare tra il valore Ct del modello e il logaritmo del numero di copie iniziale del modello.Maggiore è la concentrazione iniziale del templato, minore è il valore Ct;minore è la concentrazione iniziale del templato, maggiore è il valore Ct.
2. Curva di amplificazione, soglia di fluorescenza e certa quantità di prodotto PCR
La quantità di prodotto di amplificazione qPCR viene presentata direttamente sotto forma di segnale fluorescente, ovvero la curva di amplificazione.Nella fase iniziale della PCR, l'amplificazione è in condizioni ideali, il numero di cicli è ridotto, l'accumulo di prodotto è ridotto e il livello di fluorescenza non può essere chiaramente distinto dallo sfondo di fluorescenza.Successivamente, la fluorescenza aumenta ed entra nella fase esponenziale.La quantità di prodotto PCR può essere rilevata a un certo punto quando la reazione PCR è solo nella fase esponenziale, che può essere utilizzata come "una certa quantità di prodotto", e da ciò si può dedurre il contenuto iniziale del templato.Pertanto, l'intensità del segnale di fluorescenza corrispondente a una certa quantità di prodotto è la soglia di fluorescenza.
Nella fase avanzata della PCR, la curva di amplificazione non mostra più l'amplificazione esponenziale ed entra nella fase lineare e nella fase di plateau.
3. Riproducibilità dei valori Ct
Quando il ciclo PCR raggiunge il numero di ciclo del valore Ct, è appena entrato nel vero periodo di amplificazione esponenziale.In questo momento, il piccolo errore non è stato amplificato, quindi la riproducibilità del valore Ct è eccellente, ovvero lo stesso templato viene amplificato in momenti diversi o in provette diverse contemporaneamente.Amplificazione, il valore Ct ottenuto è costante.
1. Efficienza di amplificazione En
L'efficienza di amplificazione della PCR si riferisce all'efficienza con cui la polimerasi converte il gene da amplificare in un amplicone.L'efficienza di amplificazione quando una molecola di DNA viene trasformata in due molecole di DNA è del 100%.L'efficienza di amplificazione è comunemente espressa come En.Per facilitare l'analisi degli articoli successivi, vengono brevemente introdotti i fattori che influenzano l'efficienza dell'amplificazione.
| Fattori influenzanti | spiegazione | Come giudicare? |
| A. Inibitori della PCR | 1. Il DNA stampo contiene sostanze che inibiscono la reazione PCR, come proteine o detergenti.2. Il cDNA dopo la trascrizione inversa contiene un'alta concentrazione di componenti del reagente RNA stampo o RT, che possono anche inibire la successiva reazione PCR. | 1. La presenza di inquinamento può essere valutata misurando il rapporto tra A260/A280 e A260/A230 o l'elettroforesi dell'RNA.2. Se il cDNA è diluito secondo un certo rapporto dopo la trascrizione inversa. |
| B. Design improprio del primer | I primer non si temprano in modo efficiente | Controlla i primer per primer-dimeri o forcine, mancate corrispondenze e talvolta si estendono su design intronici. |
| C. Progettazione impropria del programma di reazione PCR | 1. I primer non possono essere ricotti efficacemente2. Rilascio insufficiente di DNA polimerasi 3. L'attività della DNA polimerasi ad alta temperatura a lungo termine è diminuita | 1. La temperatura di ricottura è superiore al valore TM del primer2. Il tempo di pre-denaturazione è troppo breve 3. Il tempo di ciascuna fase della procedura di reazione è troppo lungo |
| D. Miscelazione insufficiente dei reagenti o errori di pipettaggio | Nel sistema di reazione, la concentrazione locale dei componenti della reazione PCR è troppo alta o irregolare, con conseguente amplificazione non esponenziale dell'amplificazione PCR | |
| E. Lunghezza dell'amplicone | La lunghezza dell'amplicone è troppo lunga, superiore a 300 bp, e l'efficienza di amplificazione è bassa | Verificare che la lunghezza dell'amplicone sia compresa tra 80 e 300 bp |
| F. Influenza dei reagenti qPCR | La concentrazione di DNA polimerasi nel reagente è bassa o la concentrazione di ioni nel tampone non è ottimizzata, con conseguente attività dell'enzima Taq che non raggiunge il massimo | Determinazione dell'efficienza di amplificazione mediante curva standard |
2.Intervallo di valori Ct
I valori Ct vanno da 15 a 35.Se il valore Ct è inferiore a 15, si considera che l'amplificazione rientri nell'intervallo del periodo di riferimento e che la soglia di fluorescenza non sia stata raggiunta.Idealmente, esiste una relazione lineare tra il valore Ct e il logaritmo del numero di copie iniziale del modello, ovvero la curva standard.Attraverso la curva standard, quando l'efficienza di amplificazione è del 100%, il valore Ct calcolato per quantificare il numero di singola copia del gene è di circa 35. Se è maggiore di 35, il numero di copia iniziale del modello è teoricamente inferiore a 1, che può essere considerato privo di significato.
Per diversi intervalli Ct del gene, a causa della differenza nel numero di copie geniche e nell'efficienza di amplificazione nella quantità iniziale del templato, è necessario creare una curva standard per il gene e calcolare l'intervallo di rilevamento lineare del gene.
3.Fattori che influenzano il valore Ct
Dalla relazione tra il numero di cicli di amplificazione e la quantità di prodotto: quantità di prodotto amplificato = quantità di templato iniziale × (1+En) numero di ciclo, si può vedere che in condizioni ideali, la quantità di templato iniziale e En avrà un impatto negativo sul valore Ct.La differenza nella qualità del templato o nell'efficienza dell'amplificazione farà sì che il valore Ct sia troppo grande o troppo piccolo.
4.Il valore Ct è troppo grande o troppo piccolo
Tempo di pubblicazione: 22 febbraio 2023