PCR, multiplo PCR, PCR in situ, PCR inversa, RT-PCR, qPCR(1)–PCR
Analizzeremo i concetti, i passaggi e i dettagli di varie PCR
Ⅰ. PCR
La reazione a catena della polimerasi, denominata PCR, è una tecnologia biologica molecolare che viene utilizzata per ingrandire specifici frammenti di DNA.Può essere considerato come una speciale replicazione del DNA in vitro.La DNA polimerasi (DNA polimerasi I) è stata scoperta già nel 1955 e il frammento di Klenow di E. Coli, che ha valore sperimentale e praticità, è stato scoperto dal Dr. H. Klenow all'inizio degli anni '70, ma poiché questo enzima non tollera la temperatura, l'alta temperatura può degenerarlo, quindi non incontra la reazione a catena della polimerasi con degenerazione ad alta temperatura.Gli enzimi oggi in uso (chiamati Taq polimerasi), sono stati isolati da Thermus aquaticus, un batterio termale nel 1976. La sua caratteristica è che può resistere alle alte temperature ed è un enzima ideale, ma è ampiamente utilizzato dopo gli anni '80.Il concetto originale del prototipo primitivo originale della PCR è simile alla riparazione e copiatura genica, proposta dal Dr. KJell Kleppe nel 1971. Ha pubblicato la prima copia genica semplice ea breve termine (simile alle prime due reazioni cicliche della PCR).La PCR sviluppata oggi è stata sviluppata dal Dr. Kary B. Mullis nel 1983. Il Dr. Mullis ha servito le aziende di PE quell'anno, quindi il PE ha uno status speciale nel settore della PCR.Il Dr. Mullis ha pubblicato ufficialmente il primo documento correlato con Saiki e altri nel 1985. Da allora, l'uso della PCR è di migliaia di chilometri al giorno e si può dire che la qualità dei documenti correlati rende sgradevoli molti altri metodi di ricerca.Successivamente, la tecnologia PCR è ampiamente utilizzata nella ricerca scientifica biologica e nelle applicazioni cliniche, diventando la tecnologia più importante della ricerca in biologia molecolare.Mullis vinse anche il Premio Nobel per la Chimica nel 1993.
PCRPrincipio
Il principio di base della tecnologia PCR è simile al processo di replicazione naturale del DNA e la sua specificità dipende dal primer oligonucleotidico che è complementare a entrambe le estremità della sequenza target.La PCR è composta da tre fasi di reazione di base che estendono la degenerazione-annealing: ①Degenerazione del DNA modello: dopo che il DNA modello è stato riscaldato a circa 93°C per un certo periodo di tempo, la doppia soluzione di DNA per il DNA a catena doppia formata dall'amplificazione PCR del DNA modello in uscita, ne fa una singola catena in modo che possa essere combinata con il primer per prepararsi alla reazione successiva.②La ricottura (composto) del DNA modello e del primer: dopo che il DNA modello è stato riscaldato e degenerato in una singola catena, la temperatura scende a circa 55°C.La sequenza complementare del primer e del modello DNA a catena singola.③L'estensione del primer: DNA template-il legame del primer si basa sull'azione della TaqDNA polimerasi, con dNTP come materia prima di reazione.Mantenere il principio di replicazione, sintetizzare una nuova catena di copie semi-riservate che integri la catena del DNA modello e ripetere i tre processi di degenerazione-annealing-estensione del ciclo possono ottenere più "catena di copie semi-riservate" e questa nuova catena è di nuovo disponibile Diventa un modello per il ciclo successivo.Occorrono 2-4 minuti per completare il ciclo, il gene bersaglio può essere amplificato diversi milioni di volte in 2-3 ore.
StandardPCRSistema di reazione
| Taq DNA polimerasi | 2,5 microlitri |
| Mg2+ | 1,5mmol/l |
| Tampone di amplificazione 10 × | 10μl |
| 4 miscele dNTP | 200μl |
| DNA modello | 0,1~2μg |
| Primer | 10~100μl |
| Aggiungi acqua a vapore doppia o tripla | 100 microlitri |
Cinque elementi della reazione PCR
Ci sono principalmente cinque tipi di sostanze coinvolte nella reazione PCR, vale a dire primer, enzima, dNTP, stampo e tampone (è richiesto Mg2+).[Procedura PCR]
Il processo PCR standard è suddiviso in tre fasi
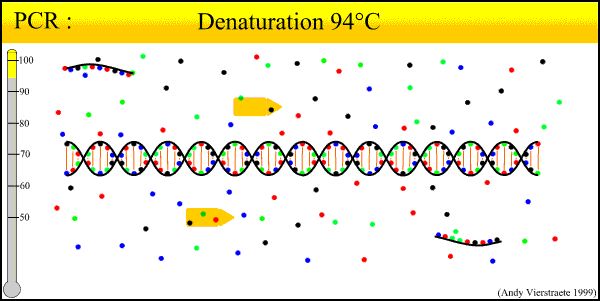
1. Degenerazione del DNA (90°C-96°C): modelli di DNA a doppia catena sotto azione termica, i legami idrogeno si rompono, formando un DNA a catena singola.
2. Ricottura (25℃ -65℃): la temperatura del sistema viene ridotta, il primer viene combinato con il modello di DNA per formare una doppia catena locale.
3. Estensione (70 ℃ -75 ℃): sotto l'azione dell'enzima Taq (circa 72 ° C, la migliore attività), dNTP viene utilizzato come materia prima, si estende dall'estremità 5 'del primer → 3' fine, la sintesi e il modello si completano a vicenda catena di DNA.
Ogni ciclo viene denaturato, ricotto ed esteso, raddoppiando il contenuto di DNA.Allo stato attuale, a causa della breve area di amplificazione, alcune PCR possono essere replicate in un tempo molto breve anche se l'attività dell'enzima Taq non è ottimale, quindi può essere modificata in due passaggi, ovvero la ricottura e l'estensione possono essere eseguite a 60°C-65°C contemporaneamente.Al fine di ridurre il processo di sollevamento e raffreddamento e migliorare la velocità di risposta.
Caratteristiche della reazione PCR
● Elevata specificità
I fattori decisivi specifici della risposta PCR sono: ①La combinazione specifica del primer e del DNA stampo.②Il principio dell'accoppiamento delle basi.③La lealtà della reazione di sintesi della TaqDNA polimerasi.④Specificità e conservatività del gene bersaglio.
La corretta combinazione di primer e modelli è la chiave.Il legame del primer e del templato e l'estensione della catena del primer si basano sul principio dell'adattamento della base alcalina.La lealtà delle reazioni di sintesi della polimerasi e la resistenza alle alte temperature della Taq DNA polimerasi per creare il legame (composto) del modello e del primer nella reazione possono essere eseguite a una temperatura più elevata.La specificità della combinazione è notevolmente aumentata.La clip può mantenere un alto grado di correttezza.Selezionando una regione genetica target con elevata conservatività e alta conservatività, la sua specificità è maggiore.
● Alta sensibilità
Il volume di produzione dei prodotti PCR è aumentato dall'indice, che può espandere il modello iniziale di Picker (PG=10-12) per aumentare il livello del microcontrollore al livello di microgrammi (μg= -6).Una cellula bersaglio può essere rilevata da 1 milione di cellule;nella rilevazione dei virus, la sensibilità della PCR può raggiungere 3 RFU (unità formate da punti vuoti);il tasso minimo di rilevamento nella scienza batterica è di 3 batteri.
● Semplice e veloce
La riflessione PCR utilizza una Taq DNA polimerasi ad alta temperatura, che aggiunge la soluzione di reazione in una sola volta, ovvero una reazione di degenerazione-anneal-estensione sulla soluzione di amplificazione del DNA e sul recipiente del bagno d'acqua.Generalmente, la reazione di amplificazione viene completata in 2-4 ore.I prodotti aumentati vengono generalmente analizzati con una spada elettrica e non devono utilizzare isotopi, nessun inquinamento radioattivo e una facile promozione.
● La purezza del campione è bassa
Non è necessario separare virus o batteri e cellule di coltura.I prodotti grezzi del DNA e l'RNA possono essere usati come amplificatori.Il rilevamento dell'amplificazione del DNA può essere utilizzato direttamente utilizzando campioni clinici come sangue, liquido corporeo, liquido per lavare la tosse, capelli, cellule e tessuto vivente.
PCRproblemi comuni
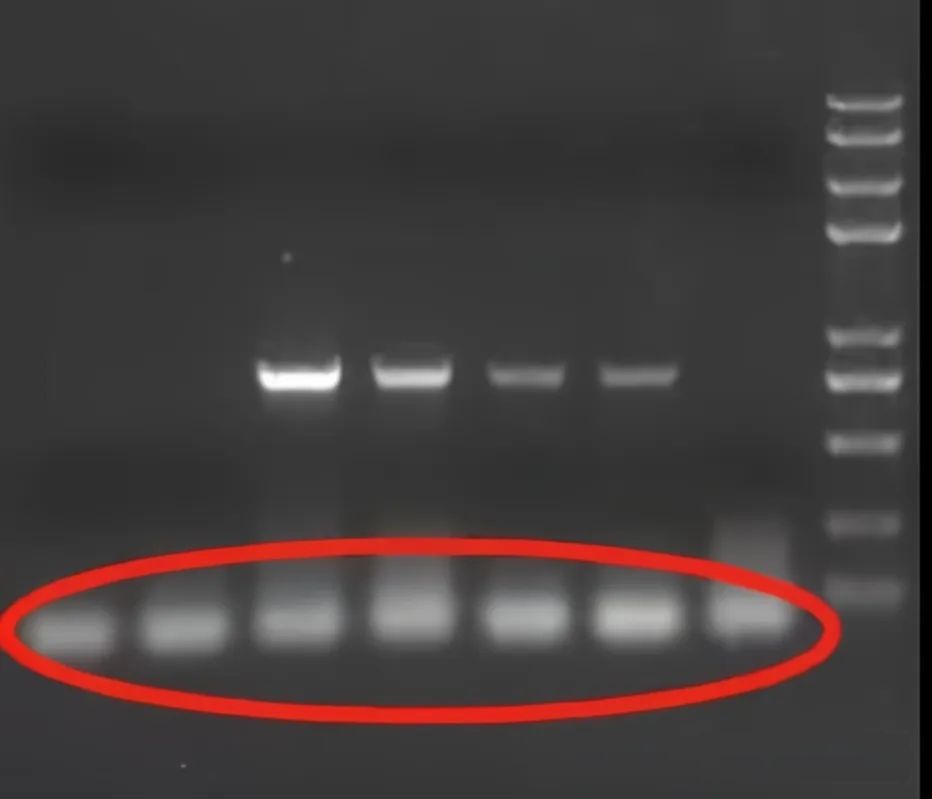
● Falso negativo, nessuna banda amplificata
Le fasi chiave della reazione PCR includono: ① preparazione degli acidi nucleici modello, ② qualità e specificità dei primer, ③ qualità degli enzimi ④ condizioni del ciclo PCR.Trovare il motivo dovrebbe essere analizzato e studiato anche per i collegamenti di cui sopra.
Modelli: ① Il modello contiene proteine varie, ② Il modello contiene un inibitore dell'enzima Taq, ③ La proteina nel modello non viene eliminata, in particolare la proteina di gruppo nel cromosoma.⑤ Deminer degenerazione dell'acido nucleico non è completa.Quando la qualità degli enzimi e dei primer è buona, non c'è banda di amplificazione, che è molto probabilmente il trattamento digestivo dei campioni.C'è qualcosa che non va nel processo di estrazione dell'acido nucleico modello, quindi per preparare una soluzione di digestione efficace e stabile, la sua procedura dovrebbe essere fissata e non modificata arbitrariamente.
Inattivazione enzimatica: un nuovo enzima o entrambi gli enzimi vecchi e nuovi dovrebbero essere usati insieme per analizzare se l'attività enzimatica è persa o insufficiente, portando a falsi negativi.Va notato che l'enzima Taq o il bromuro di etidio a volte viene dimenticato.
Primer: la qualità del primer, la concentrazione del primer e se la concentrazione dei due primer è simmetrica.È un motivo comune per il fallimento della PCR o la banda in aumento non è l'ideale e tende a diffondersi.Ci sono problemi con la qualità dei primer di alcuni numeri di lotto.I due primer hanno un'alta concentrazione e una bassa concentrazione, causando un'amplificazione asimmetrica a bassa efficienza.Le contromisure sono: ① Selezionare un buon primer per sintetizzare le unità.② La concentrazione del primer non dipende solo dal valore OD, ma presta anche attenzione al liquido originale del primer per eseguire l'elettroforesi su gel di zucchero di agar.Deve esserci una zona della striscia di primer e la luminosità dei due primer deve essere generalmente coerente.Belt, la PCR potrebbe fallire in questo momento e dovrebbe essere risolta con l'unità di sintesi dei primer.Se un primer è alto, la luminosità è bassa e la sua concentrazione deve essere bilanciata quando viene diluito.③ Il primer deve essere versato e conservato ad alta concentrazione per evitare il congelamento multiplo o parti di refrigerazione a lungo termine del frigorifero, che causeranno il deterioramento e il degrado del primer.④ Il design del primer è irragionevole, ad esempio la lunghezza del primer è insufficiente e il di cluster si forma tra i primer.
Mg2+concentrazione: la concentrazione di ioni Mg2+ ha un grande impatto sull'efficienza dell'amplificazione della PCR.Una concentrazione eccessiva può ridurre il sesso opposto dell'amplificazione PCR.Se la concentrazione è troppo bassa, l'uscita dell'amplificazione della PCR provocherà persino un fallimento dell'amplificazione della PCR senza la banda di espansione.
Modifica del volume di reazione: il volume utilizzato nell'amplificazione PCR è 20ul, 30ul e 50ul o 100uL, l'ampio volume dell'applicazione per l'amplificazione PCR è impostato in base ai diversi scopi della ricerca scientifica e dei test clinici.Dopo aver realizzato piccoli volumi come 20ul, è necessario creare una condizione del cavo quando si effettua la dimensione, altrimenti fallirà.
Motivi fisici: la trasformazione è molto importante per l'amplificazione PCR.Se la temperatura di degenerazione è bassa, il tempo di degenerazione è breve, è probabile che si verifichino falsi negativi;una temperatura di ricottura troppo bassa può causare un'amplificazione non specifica e ridurre l'efficienza dell'amplificazione specifica.Influiscono fortemente sulla combinazione di primer e modelli per ridurre l'efficienza dell'amplificazione della PCR.A volte è necessario utilizzare termometri standard per rilevare la variabilità, la ricottura e la temperatura estesa nell'estensione o nel fornello solubile in acqua, che è uno dei motivi del fallimento della PCR.
Varianti della sequenza target: se si verifica la sequenza target, una mutazione o delezione, la combinazione del prototipo e del modello è combinata, o a causa della mancanza di una sequenza target, il primer e il modello perderanno la sequenza complementare e la sua amplificazione PCR non avrà successo.
● Falso positivo
La banda di amplificazione PCR appare coerente con la banda della sequenza target e talvolta la sua banda è più precisa e più alta.
Il design del primer non è appropriato: la sequenza di amplificazione selezionata e la sequenza di amplificazione non mirata sono omologhe, quindi durante l'amplificazione PCR, i prodotti PCR amplificati sono sequenze non mirate.La sequenza target è troppo corta o il primer è troppo corto ed è soggetto a falsi positivi.Bisogno di essere riprogettato.
Inquinamento incrociato della sequenza bersaglio o dei prodotti di amplificazione: ci sono due ragioni per questo inquinamento: primo, inquinamento incrociato dell'intero genoma o di ampi segmenti, che porta a falsi positivi.Questo tipo di falso positivo può essere risolto con i seguenti metodi: Prestare attenzione e delicatezza durante il funzionamento per evitare che la sequenza target venga inalata nella pistola di campionamento o schizzi fuori dal tubo centrifugo.Fatta eccezione per gli enzimi e le sostanze che non possono resistere alle alte temperature, tutti i reagenti o le apparecchiature devono essere disinfettati ad alta pressione.I tubi ei campioni centrifughi devono essere utilizzati contemporaneamente.Se necessario, prima di aggiungere i campioni, la provetta di reazione e il reagente vengono esposti ai raggi ultravioletti per distruggere l'acido nucleico esistente.In secondo luogo, piccoli frammenti nell'inquinamento atmosferico.Questi piccoli frammenti sono più corti della sequenza bersaglio, ma hanno una certa omologia.Può essere unito l'uno con l'altro.Dopo aver completato i primer, il prodotto PCR può essere espanso, il che causerà una produzione di falsi positivi.Può essere utilizzato per ridurre o eliminare il metodo nest PCR.
● Compaiono bande di amplificazione non specifiche
Le bande che sono apparse dopo l'amplificazione PCR non sono coerenti con le dimensioni previste, o grandi o piccole, o allo stesso tempo, o allo stesso tempo, bande di amplificazione specifiche e bande di amplificazione non specifiche.L'emergere di bande non specifiche è: in primo luogo, i primer sono complementari incompleti alla sequenza target, o la polimerizzazione del primer per formare un di cluster.Il secondo è che la concentrazione di ioni MG2+ è troppo alta, la temperatura di ricottura è troppo bassa e il numero di cicli di PCR è correlato.In secondo luogo, la qualità e la quantità di enzimi.Spesso, gli enzimi di alcune fonti sono inclini a bande non speciali e gli enzimi dell'altra fonte non si verificano.A volte si verifica anche un'amplificazione non specifica degli enzimi.Le contromisure sono: attrattive ridisegnate se necessario.Ridurre la quantità di enzima o sostituire l'enzima di un'altra fonte.Ridurre la quantità di primario, aumentare la quantità di modelli in modo appropriato e ridurre il numero di cicli.Aumentare correttamente la temperatura di ricottura o utilizzare il metodo a due punti di temperatura (degenerazione a 93°C, ricottura ed estensione a circa 65°C).
● Apparire traballante traino o nastro adesivo
L'amplificazione PCR a volte sembra essere applicata o sgusciata o cintura simile a un tappeto.Per il motivo, a causa dell'eccessiva quantità di enzimi o della scarsa qualità dell'enzima, la concentrazione di dNTP è troppo alta, la concentrazione di Mg2+ è troppo alta, la temperatura di ricottura è troppo bassa e il numero di cicli è eccessivo.Le contromisure sono: ①Ridurre la quantità di enzimi o cambiare l'enzima di un'altra fonte.②Ridurre la concentrazione di dNTP ③Ridurre adeguatamente la concentrazione di Mg2+.④Aumentare la quantità di modelli e ridurre il numero di cicli.
prodotti correlati
◮ Maggiore fedeltà: 6 volte quella dell'enzima Taq ordinario;
◮ Maggiore velocità di amplificazione
◮ Maggiore adattabilità del modello
◮ Maggiore efficienza di amplificazione
◮ La tolleranza ambientale è più forte: posto a 37°C per una settimana, mantenendo oltre il 90% di attività;
◮ Ha attività 5'→3' DNA polimerasi e attività 5'→3' esonucleasi, senza attività 3'→5' esonucleasi.
L'esclusivo sistema di reazione e la Taq DNA polimerasi ad alta efficienza fanno sì che la reazione PCR abbia una maggiore efficienza di amplificazione, specificità e sensibilità.
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
◮ Il kit one-step rende la trascrizione inversa e qPCR due reazioni nella stessa provetta, è sufficiente aggiungere RNA modello, primer PCR specifici e ddH RNase-Free2O.
◮ Il kit può analizzare quantitativamente in modo rapido ed efficiente l'RNA virale o tracciare l'RNA.
◮ Il kit utilizza un esclusivo reagente di trascrizione inversa Foregene e Foregene HotStar Taq DNA Polymerase combinati con un sistema di reazione unico per migliorare efficacemente l'efficienza di amplificazione e la specificità della reazione.
◮ Il sistema di reazione ottimizzato fa sì che la reazione abbia una maggiore sensibilità di rilevamento, una maggiore stabilità termica e una migliore tolleranza.
◮ RT-qPCR FacileTMIl kit (One Step)-SYBR Green I viene fornito con il colorante di riferimento interno ROX, che può essere utilizzato per eliminare lo sfondo del segnale e gli errori di segnale tra i pozzetti, il che è conveniente per i clienti da utilizzare in diversi modelli di strumenti PCR quantitativi.
RT FacileTMII (Master Premix per sintesi del cDNA del primo filamento perPCR in tempo reale)
-Efficiente capacità di rimuovere il gDNA, che può rimuovere il gDNA nel modello entro 2 minuti.
-Efficiente sistema di trascrizione inversa, bastano solo 15 minuti per completare la sintesi del cDNA del primo filamento.
-Modelli complessi: i modelli con alto contenuto di GC e struttura secondaria complessa possono anche essere invertiti con alta efficienza.
-Sistema di trascrizione inversa ad alta sensibilità, i modelli a livello di pg possono anche ottenere cDNA di alta qualità.
-Il sistema di trascrizione inversa ha un'elevata stabilità termica, la temperatura di reazione ottimale è di 42 ℃ e ha ancora buone prestazioni di trascrizione inversa a 50 ℃.
Tempo di pubblicazione: 18 marzo 2023