一、Aumentare la sensibilità del sistema di reazione:
1. Isolare l'RNA di alta qualità:
La sintesi di cDNA di successo proviene da RNA di alta qualità.L'RNA di alta qualità dovrebbe essere almeno a lunghezza intera e privo di inibitori della trascrittasi inversa come EDTA o SDS.La qualità dell'RNA determina la quantità massima di informazioni sulla sequenza che è possibile trascrivere in cDNA.Un metodo comune di purificazione dell'RNA è un metodo in un'unica fase che utilizza isotiocianato di guanidina/fenolo acido.Per prevenire la contaminazione da tracce di RNasi, l'RNA isolato da campioni ricchi di RNasi (come il pancreas) deve essere conservato in formaldeide per preservare l'RNA di alta qualità, soprattutto per la conservazione a lungo termine.L'RNA estratto dal fegato di ratto è stato sostanzialmente degradato dopo essere stato conservato in acqua per una settimana, mentre l'RNA estratto dalla milza di ratto è rimasto stabile dopo essere stato conservato in acqua per 3 anni.Inoltre, le trascrizioni più lunghe di 4 kb sono più sensibili alla degradazione da parte di RNasi in tracce rispetto alle piccole trascrizioni.Per aumentare la stabilità dei campioni di RNA conservati, l'RNA può essere sciolto in formammide deionizzata e conservato a -70°C.La formammide utilizzata per preservare l'RNA deve essere priva di detriti che degradano l'RNA.L'RNA del pancreas può essere conservato in formammide per almeno un anno.Quando ci si prepara a utilizzare l'RNA, è possibile utilizzare il seguente metodo per precipitare l'RNA: aggiungere NaCl a 0,2 M e 4 volte il volume di etanolo, porre a temperatura ambiente per 3-5 minuti e centrifugare a 10.000 × g per 5 minuti.
2. Utilizzare la trascrittasi inversa RNaseH-inattiva (RNaseH-):
Gli inibitori dell'RNasi vengono spesso aggiunti alle reazioni di trascrizione inversa per aumentare la lunghezza e la resa della sintesi del cDNA.Gli inibitori dell'RNasi devono essere aggiunti durante la reazione di sintesi del primo filamento in presenza di un tampone e di un agente riducente (come il DTT), poiché il processo precedente alla sintesi del cDNA denatura l'inibitore, rilasciando così l'RNasi legata che può degradare l'RNA.Gli inibitori della proteina RNasi prevengono solo la degradazione dell'RNA da parte dell'RNasi A, B, C e non impediscono l'RNasi sulla pelle, quindi fai attenzione a non introdurre l'RNasi dalle dita nonostante l'uso di questi inibitori.
La trascrittasi inversa catalizza la conversione dell'RNA in cDNA.Sia M-MLV che AMV hanno attività RNaseH endogena oltre alla propria attività polimerasica.L'attività dell'RNaseH e l'attività della polimerasi competono tra loro per il filamento ibrido formato tra lo stampo di RNA e il primer del DNA o il filamento di estensione del cDNA e degradano il filamento di RNA nel complesso RNA:DNA.Il modello di RNA degradato dall'attività RNaseH non può più servire come substrato efficace per la sintesi di cDNA, il che riduce la resa e la durata della sintesi di cDNA.Pertanto, sarebbe utile eliminare o ridurre notevolmente l'attività RNasiH della trascrittasi inversa.
La trascrittasi inversa SuperScript Ⅱ, la trascrittasi inversa RNaseH- MMLV e la trascrittasi inversa thermoScript, RNaseH- AMV, possono ottenere più quantità e più cDNA a lunghezza intera rispetto a MMLV e AMV.La sensibilità RT-PCR sarà influenzata dalla quantità di sintesi di cDNA.ThermoScript è molto più sensibile di AMV.La dimensione dei prodotti RT-PCR è limitata dalla capacità della trascrittasi inversa di sintetizzare cDNA, specialmente durante la clonazione di cDNA più grandi.Rispetto a MMLV, SuperScripⅡ ha aumentato significativamente la resa dei prodotti RT-PCR lunghi.La trascrittasi inversa RNaseH ha anche una maggiore termostabilità, quindi la reazione può essere eseguita a temperature superiori ai normali 37-42°C.Nelle condizioni di sintesi suggerite, utilizzare il primer oligo(dT) e 10 μCi di [α-P]dCTP.La resa totale del primo filamento è stata calcolata utilizzando il metodo di precipitazione TCA.Il cDNA a lunghezza intera è stato analizzato utilizzando bande ordinate per dimensione asportate e contate su un gel di agarosio alcalino.
3. Aumentare la temperatura di incubazione per la trascrizione inversa:
Una temperatura di incubazione più elevata aiuta ad aprire la struttura secondaria dell'RNA, aumentando la resa della reazione.Per la maggior parte dei modelli di RNA, l'incubazione dell'RNA e dei primer a 65°C senza tampone o sale, seguita da un rapido raffreddamento su ghiaccio, eliminerà la maggior parte delle strutture secondarie e consentirà ai primer di legarsi.Tuttavia, alcuni modelli hanno ancora strutture secondarie, anche dopo la denaturazione termica.L'amplificazione di questi modelli difficili può essere eseguita utilizzando la trascrittasi inversa ThermoScript e posizionando la reazione di trascrizione inversa a una temperatura più elevata per migliorare l'amplificazione.Anche temperature di incubazione più elevate possono aumentare la specificità, specialmente quando si utilizzano primer gene-specifici (GSP) per la sintesi del cDNA (vedi Capitolo 3).Se si utilizza GSP, assicurarsi che la Tm dei primer sia la stessa della temperatura di incubazione prevista.Non utilizzare oligo(dT) e primer casuali a temperature superiori a 60°C.I primer casuali richiedono un'incubazione a 25°C per 10 minuti prima di aumentare a 60°C.Oltre a utilizzare una temperatura di trascrizione inversa più elevata, la specificità può essere migliorata anche trasferendo direttamente la miscela RNA/primer dalla temperatura di denaturazione di 65°C alla temperatura di incubazione della trascrizione inversa e aggiungendo una miscela di reazione 2× preriscaldata (sintesi cDNA hot-start).Questo approccio aiuta a prevenire l'accoppiamento di basi intermolecolari che si verifica a temperature più basse.La commutazione di temperatura multipla richiesta per RT-PCR può essere semplificata utilizzando un termociclatore.
La Tth polimerasi termostabile agisce come DNA polimerasi in presenza di Mg2+ e come RNA polimerasi in presenza di Mn2+.Può essere mantenuto caldo ad una temperatura massima di 65°C.Tuttavia, la presenza di Mn2+ durante la PCR riduce la fedeltà, il che rende la Tth polimerasi meno adatta per l'amplificazione ad alta precisione, come la clonazione del cDNA.Inoltre, Tth ha una bassa efficienza di trascrizione inversa, che riduce la sensibilità e, poiché la trascrizione inversa e la PCR possono essere eseguite con un singolo enzima, le reazioni di controllo senza trascrizione inversa non possono essere utilizzate per confrontare i prodotti di amplificazione del cDNA con il DNA genomico contaminante.I prodotti di amplificazione sono stati separati.
4. Additivi che promuovono la trascrizione inversa:
Additivi tra cui glicerolo e DMSO vengono aggiunti alla reazione di sintesi del primo filamento, che può ridurre la stabilità del doppio filamento dell'acido nucleico e sciogliere la struttura secondaria dell'RNA.È possibile aggiungere fino al 20% di glicerolo o 10% di DMSO senza influire sull'attività di SuperScript II o MMLV.AMV può anche tollerare fino al 20% di glicerolo senza perdita di attività.Per massimizzare la sensibilità della RT-PCR nella reazione di trascrizione inversa SuperScriptⅡ, è possibile aggiungere glicerolo al 10% e incubato a 45°C.Se 1/10 del prodotto della reazione di trascrizione inversa viene aggiunto alla PCR, la concentrazione di glicerolo nella reazione di amplificazione è dello 0,4%, il che non è sufficiente per inibire la PCR.
5. Trattamento RNaseH:
Il trattamento delle reazioni di sintesi del cDNA con RNaseH prima della PCR può aumentare la sensibilità.Per alcuni modelli, si ritiene che l'RNA nella reazione di sintesi del cDNA impedisca il legame dei prodotti di amplificazione, nel qual caso il trattamento con RNaseH può aumentare la sensibilità.Generalmente, il trattamento con RNaseH è necessario quando si amplificano modelli target di cDNA a lunghezza intera più lunghi, come la scherosi tuberosa II a bassa copia.Per questo modello difficile, il trattamento con RNaseH ha migliorato il segnale prodotto da SuperScript II o cDNA sintetizzato da AMV.Per la maggior parte delle reazioni RT-PCR, il trattamento con RNaseH è facoltativo, poiché la fase di denaturazione della PCR a 95°C generalmente idrolizza l'RNA nel complesso RNA:DNA.
6. Miglioramento del metodo di rilevamento del piccolo RNA:
RT-PCR è particolarmente impegnativo quando sono disponibili solo piccole quantità di RNA.Il glicogeno aggiunto come vettore durante l'isolamento dell'RNA aiuta ad aumentare la resa di piccoli campioni.Il glicogeno privo di RNasi può essere aggiunto contemporaneamente all'aggiunta di Trizol.Il glicogeno è solubile in acqua e può essere mantenuto nella fase acquosa con l'RNA per favorire la successiva precipitazione.Per campioni inferiori a 50 mg di tessuto o 106 cellule in coltura, la concentrazione raccomandata di glicogeno privo di RNasi è di 250 μg/ml.
L'aggiunta di BSA acetilata alla reazione di trascrizione inversa utilizzando SuperScript II può aumentare la sensibilità e, per piccole quantità di RNA, ridurre la quantità di SuperScript II e aggiungere 40 unità di inibitore della nucleasi RNaseOut può aumentare il livello di rilevamento.Se il glicogeno viene utilizzato nel processo di isolamento dell'RNA, si consiglia comunque di aggiungere l'inibitore della BSA o dell'RNasi quando si utilizza SuperScript II per la reazione di trascrizione inversa.
二、Aumentare la specificità RT-PCR
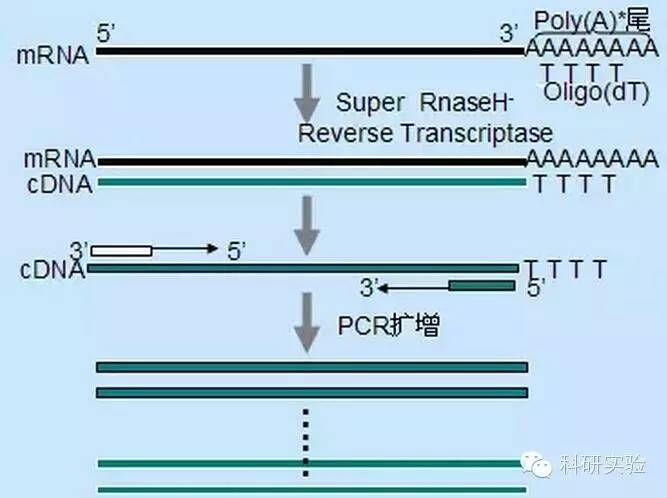
1. Asintesi CND:
La sintesi del cDNA del primo filamento può essere avviata utilizzando tre diversi metodi, la cui specificità relativa influenza la quantità e il tipo di cDNA sintetizzato.
Il metodo del primer casuale era il meno specifico dei tre metodi.I primer si ricottura in più siti durante la trascrizione, generando cDNA corti e di lunghezza parziale.Questo metodo è frequentemente utilizzato per ottenere sequenze terminali 5' e per ottenere cDNA da stampi di RNA con regioni di struttura secondaria o con siti di terminazione che non possono essere replicati dalla trascrittasi inversa.Per ottenere il cDNA più lungo, il rapporto tra primer e RNA in ciascun campione di RNA deve essere determinato empiricamente.La concentrazione iniziale di primer casuali variava da 50 a 250 ng per 20 μl di reazione.Poiché il cDNA sintetizzato dall'RNA totale utilizzando primer casuali è principalmente RNA ribosomiale, il poli(A)+RNA viene generalmente scelto come stampo.
I primer Oligo(dT) sono più specifici dei primer casuali.Si ibrida alla coda poli (A) che si trova all'estremità 3 'della maggior parte degli mRNA eucariotici.Poiché il poli(A)+ RNA è circa dall'1% al 2% dell'RNA totale, la quantità e la complessità del cDNA è molto inferiore rispetto ai primer casuali.A causa della sua elevata specificità, l'oligo(dT) generalmente non richiede l'ottimizzazione del rapporto tra RNA e primer e la selezione di poli(A)+.Si consiglia di utilizzare 0,5 μg di oligo(dT) per sistema di reazione da 20 μl.oligo(dT)12-18 è adatto per la maggior parte delle RT-PCR.Il sistema ThermoScript RT-PCR offre oligo(dT)20 grazie alla sua migliore stabilità termica per temperature di incubazione più elevate.
I primer gene specifici (GSP) sono i primer più specifici per la fase di trascrizione inversa.GSP è un oligonucleotide antisenso che può ibridarsi in modo specifico alla sequenza target dell'RNA, a differenza dei primer casuali o dell'oligo(dT), che si associano a tutti gli RNA.Le stesse regole utilizzate per progettare i primer PCR si applicano alla progettazione di GSP nelle reazioni di trascrizione inversa.L'SPG può essere la stessa sequenza del primer di amplificazione che si associa all'estremità 3′ più alta dell'mRNA, oppure l'SPG può essere progettato per l'anneal a valle del primer di amplificazione inversa.Per alcuni soggetti amplificati, è necessario progettare più di un primer antisenso per una RT-PCR di successo perché la struttura secondaria dell'RNA bersaglio può impedire il legame del primer.Si consiglia di utilizzare 1 pmol antisenso GSP in una reazione di sintesi del primo filamento da 20 μl.
2. Aumentare la temperatura di incubazione per la trascrizione inversa:
Per sfruttare appieno il pieno vantaggio della specificità GSP, dovrebbe essere utilizzata una trascrittasi inversa con maggiore termostabilità.Le trascrittasi inverse termostabili possono essere incubate a temperature più elevate per aumentare la severità della reazione.Ad esempio, se un GSP si appaia a 55°C, la specificità del GSP non sarà completamente utilizzata se AMV o M-MLV viene utilizzato per la trascrizione inversa a una bassa stringenza di 37°C.Tuttavia, SuperScript II e ThermoScript possono essere fatti reagire a 50°C o più, il che eliminerà i prodotti non specifici generati a temperature inferiori.Per la massima specificità, la miscela RNA/primer può essere trasferita direttamente dalla temperatura di denaturazione di 65°C alla temperatura di incubazione della trascrizione inversa e aggiunta a una miscela di reazione 2× preriscaldata (avvio a caldo della sintesi di cDNA).Questo aiuta a prevenire l'accoppiamento di basi intermolecolari a basse temperature.Le molteplici transizioni di temperatura richieste per RT-PCR possono essere semplificate utilizzando un termociclatore.
3. Riduce la contaminazione del DNA genomico:
Una potenziale difficoltà incontrata con RT-PCR è la contaminazione del DNA genomico nell'RNA.L'utilizzo di un buon metodo di isolamento dell'RNA, come il Trizol Reagent, ridurrà la quantità di DNA genomico che contamina la preparazione dell'RNA.Per evitare prodotti derivati dal DNA genomico, l'RNA può essere trattato con DNasi I di grado di amplificazione per rimuovere il DNA contaminante prima della trascrizione inversa.La digestione della DNasi I è stata terminata incubando i campioni in EDTA 2,0 mM per 10 minuti a 65°C.L'EDTA può chelare gli ioni di magnesio, prevenendo l'idrolisi dell'RNA dipendente dagli ioni di magnesio ad alte temperature.
Al fine di separare il cDNA amplificato dai prodotti di amplificazione del DNA genomico contaminanti, è possibile progettare primer in modo che ciascuno si ricottura per separare gli esoni.I prodotti di PCR derivati dal cDNA saranno più corti di quelli derivati dal DNA genomico contaminato.Inoltre, su ciascun modello di RNA è stato eseguito un esperimento di controllo senza trascrizione inversa per determinare se un dato frammento fosse derivato da DNA genomico o cDNA.Il prodotto di PCR ottenuto senza trascrizione inversa è derivato dal genoma.
Tempo di pubblicazione: maggio-16-2023








