Ⅰ. Aumentare la sensibilità del sistema di reazione:
1. RNA separato di alta qualità:
La sintesi di cDNA di successo proviene da RNA di alta qualità.L'RNA di alta qualità dovrebbe garantire almeno un totale più lungo e non contiene inibitori che non contengono enzimi di registrazione, come EDTA o SDS.La qualità dell'RNA determina il valore massimo delle informazioni sulla sequenza che è possibile trascrivere nel cDNA.Il metodo generale di purificazione dell'RNA è un metodo graduale di utilizzo di isoocianato/acidofenolo.Al fine di prevenire l'inquinamento da RNasi, l'RNA separato da un campione ricco di RNasi (come il pancreas) richiede la conservazione di formaldeide per salvare RNA di alta qualità, il che è ancora più vero per la conservazione a lungo termine.L'RNA estratto dal fegato di ratto è stato sostanzialmente degradato dopo una settimana di conservazione in acqua, mentre l'RNA estratto dalla milza di ratto è rimasto stabile dopo tre anni di conservazione in acqua.Inoltre, i trascritti più grandi di 4 kb sono più sensibili alla traccia della degradazione dell'RNasi rispetto ai piccoli trascritti.Per aumentare la stabilità del campione di RNA di conservazione, l'RNA può essere sciolto in una metalmamina di ioni e conservato a -70 °C.La tilide utilizzata per salvare l'RNA non deve contenere un oggetto vario che degrada l'RNA.L'RNA, che deriva dal pancreas, può essere conservato nella metalmamina per almeno un anno.Quando si è pronti per utilizzare l'RNA, è possibile utilizzare i seguenti metodi per precipitare l'RNA: aggiungere NaCl a 0,2 me 4 volte il volume di etanolo, posizionare a temperatura ambiente per 3-5 minuti e centrifugare a 10.000 × g per 5 minuti.
2. Utilizzare la trascrittasi inversa senza attività RNaseH (RNaseH-):
Gli inibitori dell'RNasi vengono spesso aggiunti alle reazioni di trascrizione inversa per aumentare la lunghezza e la resa della sintesi del cDNA.L'inibitore dell'RNasi viene aggiunto nella prima reazione di sintesi della catena in presenza di tamponi e agenti riducenti come DTT perché il processo di sintesi pre-cDNA denatura l'inibitore, rilasciando così RNasi legate che degradano l'RNA.L'inibitore della proteina RNasi previene solo la degradazione dell'RNA da parte dell'RNasi A, B, C e non previene le RNasi sulla pelle, quindi è necessario prestare attenzione a non introdurre RNasi dalle dita nonostante l'uso di questi inibitori.
La trascrittasi inversa catalizza la conversione dell'RNA in cDNA.Sia M-MLV che AMV hanno attività RNaseH endogena oltre alla propria attività polimerasica.L'attività dell'RNaseH compete con l'attività della polimerasi per i filamenti eterozigoti formati tra modelli di RNA e primer di DNA o filamenti di estensione del cDNA e degrada l'RNA: filamenti di RNA nei complessi di DNA.I modelli di RNA degradati dall'attività RNaseH non possono più essere utilizzati come substrati efficaci per la sintesi di cDNA, riducendo la resa e la durata della sintesi di cDNA.Quindi eliminare o ridurre notevolmente l'attività RNaseH della trascrittasi inversa sarebbe di grande beneficio.
La trascrittasi inversa SuperScriptⅡ, la trascrittasi inversa MMLV di RNaseH- e la trascrittasi inversa thermoScript, AMV di RNaseH- hanno prodotto più cDNA a lunghezza intera rispetto a MMLV e AMV.La sensibilità RT-PCR è influenzata dalla quantità di cDNA sintetizzato.ThermoScript è molto più sensibile di AMV.La dimensione dei prodotti RT-PCR è limitata dalla capacità della trascrittasi inversa di sintetizzare cDNA, specialmente durante la clonazione di Cdna più grandi.Rispetto a MMLV, SuperScripⅡ ha aumentato significativamente la resa dei prodotti RT-PCR lunghi.La trascrittasi inversa di RNaseH- aumenta anche la stabilità termica, quindi la reazione può essere eseguita a temperature superiori al normale di 37-42 ℃.Nelle condizioni di sintesi suggerite, sono stati utilizzati primer oligo(dT) e 10μCi [alpha-p]dCTP.La produzione totale della prima catena è stata calcolata utilizzando il metodo di precipitazione TCA.Il cDNA a lunghezza intera è stato analizzato utilizzando la rimozione di strisce ordinate per dimensione e il conteggio in un gel di agarosio alcalino.
3. Aumentare la temperatura di conservazione del calore della trascrizione inversa:
Una temperatura di mantenimento più elevata aiuta ad aprire la struttura secondaria dell'RNA e ad aumentare la resa della reazione.Per la maggior parte dei modelli di RNA, mantenere l'RNA e il primer a 65°C senza tampone o sale e quindi raffreddarli rapidamente su ghiaccio elimina la maggior parte delle strutture secondarie e consente ai primer di legarsi.Tuttavia, alcuni modelli hanno ancora una struttura secondaria, anche dopo la denaturazione termica.L'amplificazione di questi modelli difficili può essere eseguita utilizzando la trascrittasi inversa ThermoScript e ponendo la reazione della trascrittasi inversa a temperature più elevate per migliorare l'amplificazione.Temperature di mantenimento più elevate possono anche aumentare la specificità, specialmente quando la sintesi del cDNA viene eseguita utilizzando primer gene-specifici (GSPS) (vedi Capitolo 3).Se si utilizza GSP, assicurarsi che il valore Tm del primer sia lo stesso della temperatura di mantenimento prevista.Non utilizzare oligo(dT) e primer casuali al di sopra di 60℃.I primer casuali devono essere mantenuti a 25 ℃ per 10 minuti prima di aumentare a 60 ℃.Oltre a utilizzare temperature di trascrizione inversa più elevate, la specificità può essere migliorata trasferendo direttamente la miscela RNA/primer dalla temperatura di denaturazione di 65 ℃ alla temperatura di mantenimento della trascrizione inversa e aggiungendo una miscela di reazione 2 × preriscaldata (sintesi di iniziazione termica del cDNA).Questo approccio aiuta a prevenire l'accoppiamento di basi intermolecolari che si verifica a temperature più basse.L'utilizzo di uno strumento PCR semplifica i numerosi interruttori di temperatura richiesti per RT-PCR.
La Tth polimerasi termostabilizzata agisce come DNA polimerasi in presenza di Mg2+ e come RNA polimerasi in presenza di Mn2+.Può trattenere il calore fino a 65 ℃.Tuttavia, la presenza di Mn2+ durante la PCR riduce la fedeltà, il che rende la Tth polimerasi meno adatta per l'amplificazione ad alta precisione, come la clonazione del cDNA.Inoltre, Tth è meno efficiente nella trascrizione inversa, il che riduce la sensibilità, e poiché un singolo enzima può eseguire trascrizione inversa e PCR, le reazioni di controllo senza trascrizione inversa non possono essere utilizzate per distinguere i prodotti amplificati di cDNA da quelli del DNA genomico contaminato.
4. Additivo che promuove la trascrizione inversa:
L'aggiunta di additivi, tra cui glicerina e DMSO, alla prima reazione di sintesi della catena può ridurre la stabilità del doppio filamento dell'acido nucleico e svolgere la struttura secondaria dell'RNA.È possibile aggiungere fino al 20% di glicerina o il 10% di DMSO senza influire sull'attività di SuperScriptⅡ o MMLV.AMV può anche tollerare fino al 20% di glicerolo senza ridurre l'attività.Per massimizzare la sensibilità della RT-PCR nella reazione di trascrizione inversa SuperScriptⅡ, è possibile aggiungere il 10% di glicerolo e isolarlo a 45℃.Se 1/10 del prodotto della reazione di retrotrascrizione viene aggiunto alla PCR, la concentrazione di glicerolo nella reazione di amplificazione è dello 0,4%, che non è sufficiente per inibire la PCR.
5. Elaborazione RNaseH:
La sensibilità può essere migliorata trattando le reazioni di sintesi del cDNA con RNaseH prima della PCR.Per alcuni modelli, si ritiene che l'RNA nella reazione di sintesi del cDNA impedisca il legame di prodotti amplificati, nel qual caso il trattamento con RNaseH può aumentare la sensibilità.Generalmente, il trattamento con RNaseH è necessario per l'amplificazione di un modello target di cDNA a lunghezza intera relativamente lungo, come la scherosi tuberosaⅡ con bassa copia.Per questo modello difficile, RNaseH ha migliorato il segnale generato dal cDNA sintetizzato da SuperScriptⅡ o AMV.Per la maggior parte delle reazioni RT-PCR, il trattamento RNaseH è facoltativo perché la fase di denaturazione PCR isolata a 95 ℃ in genere idrolizza l'RNA dal complesso RNA: DNA.
6. Metodi migliorati per rilevare piccole quantità di RNA:
RT-PCR è particolarmente impegnativo quando sono disponibili solo piccole quantità di RNA.L'aggiunta di glicogeno come vettore durante la separazione dell'RNA aiuta ad aumentare la resa di piccoli campioni.Un glicogeno privo di RNasi può essere aggiunto contemporaneamente al Trizol.Il glicogeno è solubile in acqua e può rimanere nella fase acquosa con l'RNA per favorire la successiva precipitazione.La concentrazione raccomandata di glicogeno privo di RNasi è di 250 μg/ml per campioni inferiori a 50 mg di tessuto o 106 cellule in coltura.
L'aggiunta di BSA acetilata per invertire le reazioni di trascrizione utilizzando SuperScriptⅡ può aumentare la sensibilità e, per piccole quantità di RNA, ridurre la quantità di SuperScriptⅡ e aggiungere 40 unità di inibitore della nucleasi RnaseOut può migliorare il livello di rilevamento.Se il glicogeno viene utilizzato nella separazione dell'RNA, è comunque consigliata l'aggiunta di inibitori della BSA o dell'RNasi per invertire le reazioni di trascrizione utilizzando SuperScriptⅡ.
Ⅱ. Aumentare la specificità di RT-PCR
1. Sintesi cNDA:
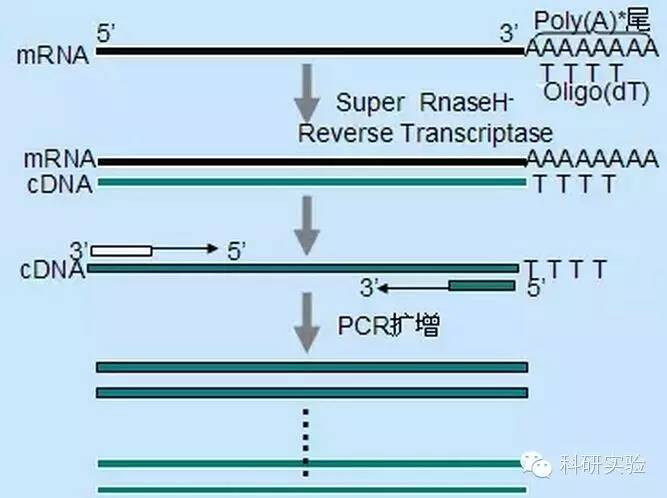
È possibile utilizzare tre diversi metodi per avviare la sintesi del cDNA del primo filamento e la specificità relativa di ciascun metodo influisce sulla quantità e sul tipo di cDNA sintetizzato.
Il metodo del primer casuale è il meno specifico dei tre metodi.I primer vengono ricotti in più siti in tutta la trascrizione per produrre cDNA corto e di lunghezza parziale.Questo metodo viene spesso utilizzato per ottenere sequenze terminali 5 'e cDNA da modelli di RNA con regioni strutturali secondarie o con siti terminali che la trascrittasi inversa non può replicare.Per ottenere il cDNA più lungo, il rapporto tra primer e RNA in ciascun campione di RNA deve essere determinato empiricamente.La concentrazione iniziale di primer casuali varia da 50 a 250 ng per sistema di reazione da 20 μl.Poiché il cDNA sintetizzato dall'RNA totale utilizzando primer casuali è principalmente RNA ribosomiale, poli(A)+RNA viene generalmente selezionato come templato.
L'inizio di Oligo(dT) è più specifico dei primer casuali.Si ibrida con la coda di poli(A) che si trova all'estremità 3′ dell'mRNA nella maggior parte delle cellule eucariotiche.Poiché il poli(A)+RNA è all'incirca dall'1% al 2% dell'RNA totale, la quantità e la complessità del cDNA è molto inferiore rispetto all'utilizzo di primer casuali.A causa della sua elevata specificità, l'oligo(dT) generalmente non richiede l'ottimizzazione per il rapporto tra RNA e primer e la selezione di poli(A)+.Si consiglia di utilizzare 0,5 μg di oligo(dT) per sistema di reazione da 20 μl.oligo(dT)12-18 è adatto per la maggior parte delle RT-PCR.Il sistema ThermoScript RT-PCR fornisce oligo(dT)20 grazie alla sua buona stabilità termica ed è adatto a temperature di mantenimento più elevate.
I primer specifici per gene (GSP) sono i migliori primer specifici per la fase di trascrizione inversa.GSP è un oligonucleoside antisenso che può specificamente ibridarsi con le sequenze di destinazione dell'RNA, piuttosto che appaiare tutti gli Rnas come primer casuali o oligo(dT).Le regole utilizzate per progettare i primer per PCR si applicano anche alla progettazione della reazione di trascrizione inversa GSP.GSP può essere la stessa sequenza del primer di amplificazione ricotto alla fine dell'mRNA3 ′, oppure GSP può essere progettato per essere ricotto a valle con il primer di amplificazione inversa.Per alcuni oggetti amplificati, è necessario progettare più di un primer antisenso per una RT-PCR di successo perché la struttura secondaria dell'RNA bersaglio può impedire il legame del primer.Si suggerisce di utilizzare 1pmol antisenso GSP nel primo sistema di reazione di sintesi a catena di 20μl.
2. Aumentare la temperatura di conservazione del calore della trascrizione inversa:
Per sfruttare appieno la specificità GSP, è necessario utilizzare la trascrittasi inversa con elevata stabilità termica.La trascrittasi inversa termostabile può essere isolata a temperature più elevate per aumentare il rigore della reazione.Ad esempio, se un GSP viene ricotto a 55°C, la specificità di GSP non viene utilizzata completamente se la trascrizione inversa viene eseguita a 37°C con basso rigore utilizzando AMV o M-MLV.Tuttavia, SuperScripⅡ e ThermoScript possono reagire a 50 ℃ o superiori, il che elimina i prodotti non specifici prodotti a temperature inferiori.Per la massima specificità, la miscela RNA/primer può essere trasferita direttamente dalla temperatura di denaturazione di 65°C alla temperatura di mantenimento della trascrizione inversa con l'aggiunta di una miscela di reazione 2x preriscaldata (inizio termico della sintesi del cDNA).Questo aiuta a prevenire l'accoppiamento di basi tra le molecole a basse temperature.L'utilizzo di uno strumento PCR semplifica le numerose transizioni di temperatura richieste per RT-PCR.
3. Ridurre la contaminazione del DNA genomico:
Una potenziale difficoltà con RT-PCR è che l'RNA contamina il DNA genomico.L'uso di migliori metodi di separazione dell'RNA, come il Trizol Reagent, riduce la contaminazione del DNA genomico nelle preparazioni di RNA.Per evitare prodotti prodotti dal DNA genomico, l'RNA può essere trattato con DnasⅠ di grado di amplificazione per rimuovere il DNA contaminato prima della trascrizione inversa.I campioni sono stati tenuti a 65 ℃ in EDTA 2,0 mM per 10 minuti per terminare la digestione con DNasiⅠ.L'EDTA chela gli ioni di magnesio per prevenire l'idrolisi dell'RNA dipendente dagli ioni di magnesio che si verifica ad alte temperature.
Per separare il cDNA amplificato dal prodotto di amplificazione del DNA del genoma, è possibile progettare primer che si appaiano separatamente con l'esone separato.I prodotti di PCR derivati dal cDNA saranno più corti di quelli derivati dal DNA genomico contaminato.Su ciascun modello di RNA viene eseguito anche un esperimento controllato senza trascrizione inversa per determinare se un dato frammento proviene da DNA genomico o cDNA.I prodotti di PCR ottenuti in assenza di trascrizione inversa derivano dal genoma.
Prodotti correlati
-Il kit one-step consente di eseguire la trascrizione inversa e la PCR nella stessa provetta.Ha solo bisogno di aggiungere template RNA, primer PCR specifici e ddH RNase-Free2O.
-L'analisi quantitativa in tempo reale dell'RNA può essere eseguita in modo rapido e preciso.
-Il kit utilizza un esclusivo reagente di trascrizione inversa Foregene e Foregene HotStar Taq DNA Polymerase combinati con un sistema di reazione unico per migliorare efficacemente l'efficienza di amplificazione e la specificità della reazione.
-Il sistema di reazione ottimizzato fa sì che la reazione abbia una maggiore sensibilità di rilevamento, una maggiore stabilità termica e una migliore tolleranza.
-Efficiente capacità di rimuovere il gDNA, che può rimuovere il gDNA nel modello entro 2 minuti.
-Efficiente sistema di trascrizione inversa, bastano solo 15 minuti per completare la sintesi del cDNA del primo filamento.
-Modelli complessi: i modelli con alto contenuto di GC e struttura secondaria complessa possono anche essere invertiti con alta efficienza.
-Sistema di trascrizione inversa ad alta sensibilità, i modelli a livello di pg possono anche ottenere cDNA di alta qualità.
-Il sistema di trascrizione inversa ha un'elevata stabilità termica, la temperatura di reazione ottimale è di 42 ℃ e ha ancora buone prestazioni di trascrizione inversa a 50 ℃.
Tempo di pubblicazione: mar-07-2023