1. Conoscenze di base (se vuoi vedere la parte sperimentale, passa direttamente alla seconda parte)
Come reazione derivata della PCR convenzionale, la PCR in tempo reale monitora principalmente la variazione della quantità di prodotto di amplificazione in ogni ciclo della reazione di amplificazione della PCR in tempo reale attraverso la variazione del segnale di fluorescenza e analizza quantitativamente il modello iniziale attraverso la relazione tra il valore ct e la curva standard.
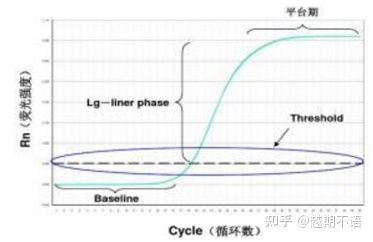
I dati specifici di RT-PCR sonolinea di base, soglia di fluorescenzaEValore CT.
| linea di base: | Il valore di fluorescenza del 3°-15° ciclo è la linea di base (linea di base), che è causata dall'errore occasionale della misurazione. |
| Soglia (soglia): | Si riferisce al limite di rilevamento della fluorescenza impostato in una posizione appropriata nella regione di crescita esponenziale della curva di amplificazione, generalmente 10 volte la deviazione standard della linea di base. |
| Valore CT: | È il numero di cicli di PCR quando il valore di fluorescenza in ciascuna provetta di reazione raggiunge la soglia. Il valore Ct è inversamente proporzionale alla quantità di templato iniziale. |
Metodi di etichettatura comuni per RT-PCR:
| metodo | vantaggio | discordanza | ambito di applicazione |
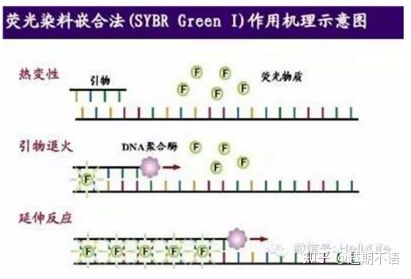
| SYBR VerdeⅠ | Ampia applicabilità, sensibile, economico e conveniente | I requisiti del primer sono elevati, inclini a bande non specifiche | È adatto per l'analisi quantitativa di vari geni bersaglio, la ricerca sull'espressione genica e la ricerca su animali e piante ricombinanti transgenici. |
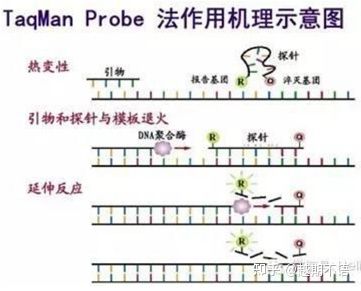
| TaqMan | Buona specificità e alta ripetibilità | Il prezzo è alto e adatto solo per obiettivi specifici. | Rilevazione di agenti patogeni, ricerca sui geni di resistenza ai farmaci, valutazione dell'efficacia dei farmaci, diagnosi delle malattie genetiche. |
| faro molecolare | Elevata specificità, fluorescenza, fondo basso | Il prezzo è alto, è adatto solo per uno scopo specifico, il design è difficile e il prezzo è alto. | Analisi genica specifica, analisi SNP |
2. Fasi sperimentali
2.1 Sul raggruppamento sperimentale- devono esserci più pozzetti nel gruppo e devono esserci ripetizioni biologiche.
| ① | Controllo vuoto | Utilizzato per rilevare lo stato di crescita cellulare negli esperimenti |
| ② | Controllo negativo siRNA (sequenza siRNA non specifica) | Dimostrare la specificità dell'azione dell'RNAi.siRNA può indurre una risposta allo stress non specifica a una concentrazione di 200 nM. |
| ③ | Controllo del reagente di trasfezione | Escludere la tossicità del reagente di trasfezione per le cellule o l'effetto sull'espressione del gene bersaglio |
| ④ | siRNA contro il gene bersaglio | Abbattere l'espressione del gene bersaglio |
| ⑤ (facoltativo) | siRNA positivo | Utilizzato per la risoluzione dei problemi del sistema sperimentale e dei problemi operativi |
| ⑥ (facoltativo) | SiRNA di controllo fluorescente | L'efficienza della trasfezione cellulare può essere osservata con un microscopio |
2.2 Principi di progettazione del primer
| Dimensione del frammento amplificata | Preferibilmente a 100-150 bp |
| Lunghezza dell'innesco | 18-25 punti base |
| Contenuto CG | 30%-70%, preferibilmente 45%-55% |
| Valore tm | 58-60℃ |
| Sequenza | Evitare T/C continuo;A/G continuo |
| 3 sequenza finale | Evitare ricchi di GC o ricchi di AT;la base terminale è preferibilmente G o C;è meglio evitare T |
| Complementarietà | Evitare sequenze complementari di più di 3 basi all'interno del primer o tra due primer |
| Specificità | Utilizzare la ricerca esplosiva per confermare la specificità del primer |
①SiRNA è specie-specifico e le sequenze di specie diverse saranno diverse.
②SiRNA è confezionato in polvere liofilizzata, che può essere conservata stabilmente per 2-4 settimane a temperatura ambiente.
2.3 Strumenti o reagenti che devono essere preparati in anticipo
| Primer (riferimento interno) | Compresi avanti e indietro due |
| Primer (gene bersaglio) | Compresi avanti e indietro due |
| Target Si RNA (3 strisce) | Generalmente, l'azienda sintetizzerà 3 strisce, quindi sceglierà una delle tre mediante RT-PCR |
| Kit di trasfezione | Lipo2000 ecc. |
| Kit di estrazione rapida dell'RNA | Per l'estrazione dell'RNA dopo la trasfezione |
| Kit di trascrizione inversa rapida | per la sintesi del cDNA |
| Kit di amplificazione PCR | 2 × Super SYBR verde Miscela master qPCR |
2.4 Per quanto riguarda le questioni a cui prestare attenzione nelle specifiche fasi sperimentali:
①processo di trasfezione dell'siRNA
1. Per la placcatura, è possibile scegliere una piastra da 24 pozzetti, una piastra da 12 pozzetti o una piastra da 6 pozzetti (la concentrazione media di RNA proposta in ciascun pozzetto di una piastra da 24 pozzetti è di circa 100-300 ng/uL) e la densità di trasfezione ottimale delle cellule è fino al 60% -80% circa
2. Le fasi di trasfezione e i requisiti specifici sono strettamente conformi alle istruzioni.
3. Dopo la trasfezione, i campioni possono essere raccolti entro 24-72 ore per il rilevamento dell'mRNA (RT-PCR) o il rilevamento delle proteine entro 48-96 ore (WB)
② Processo di estrazione dell'RNA
1. Prevenire la contaminazione da enzimi esogeni.Comprende principalmente indossare rigorosamente maschere e guanti;utilizzando puntali per pipette sterilizzati e tubi EP;l'acqua utilizzata nell'esperimento deve essere priva di RNasi.
2. Si consiglia di fare due volte come suggerito nel kit di estrazione rapida, che migliorerà davvero la purezza e la resa.
3. Il liquido di scarto non deve toccare la colonna di RNA.
③ Quantificazione dell'RNA
Dopo che l'RNA è stato estratto, può essere quantificato direttamente con Nanodrop e la lettura minima può arrivare fino a 10 ng/ul.
④Processo di trascrizione inversa
1. A causa dell'elevata sensibilità di RT-qPCR, è necessario creare almeno 3 pozzetti paralleli per ciascun campione per evitare che il Ct successivo sia troppo diverso o che la SD sia troppo grande per l'analisi statistica.
2. Non congelare e scongelare ripetutamente la Master Mix.
3. Ogni tubo/foro deve essere sostituito con una nuova punta!Non utilizzare continuamente lo stesso puntale per pipetta per aggiungere campioni!
4. La pellicola attaccata alla piastra a 96 pozzetti dopo l'aggiunta del campione deve essere lisciata con una piastra.È meglio centrifugare prima di metterlo sulla macchina, in modo che il liquido sulla parete del tubo possa scorrere verso il basso e rimuovere le bolle d'aria.
⑤ Analisi della curva comune
| Nessun periodo di crescita logaritmica | Possibilmente alta concentrazione di stampo |
| Nessun valore CT | Passaggi errati per il rilevamento di segnali fluorescenti; degradazione di primer o sonde: la sua integrità può essere rilevata mediante elettroforesi PAGE; quantità insufficiente di modello; degradazione dei modelli – evitando l'introduzione di impurità e ripetuti congelamenti e scongelamenti nella preparazione del campione; |
| CT>38 | Bassa efficienza di amplificazione;Il prodotto PCR è troppo lungo;vari componenti della reazione vengono degradati |
| Curva di amplificazione lineare | Le sonde possono essere parzialmente degradate da ripetuti cicli di congelamento-scongelamento o dall'esposizione prolungata alla luce |
| La differenza nei fori duplicati è particolarmente grande | La soluzione di reazione non è completamente fusa o la soluzione di reazione non è miscelata;il bagno termale dello strumento PCR è contaminato da sostanze fluorescenti |
2.5 Informazioni sull'analisi dei dati
L'analisi dei dati di qPCR può essere suddivisa in quantificazione relativa e quantificazione assoluta.Ad esempio, le cellule nel gruppo di trattamento confrontate con le cellule nel gruppo di controllo,
Quante volte cambia l'mRNA del gene X, questa è una quantificazione relativa;in un certo numero di cellule, l'mRNA del gene X
Quante copie ci sono, questa è una quantificazione assoluta.Di solito quello che usiamo di più in laboratorio è il metodo quantitativo relativo.Generalmente,il metodo 2-ΔΔctviene utilizzato di più negli esperimenti , quindi solo questo metodo verrà introdotto in dettaglio qui.
Metodo 2-ΔΔct: il risultato ottenuto è la differenza nell'espressione del gene bersaglio nel gruppo sperimentale rispetto al gene bersaglio nel gruppo di controllo.È necessario che le efficienze di amplificazione sia del gene target che del gene di riferimento interno siano prossime al 100% e la deviazione relativa non superi il 5%.
Il metodo di calcolo è il seguente:
Δct gruppo di controllo = valore ct del gene bersaglio nel gruppo di controllo – valore ct del gene di riferimento interno nel gruppo di controllo
Δct gruppo sperimentale = valore ct del gene bersaglio nel gruppo sperimentale – valore ct del gene di riferimento interno nel gruppo sperimentale
ΔΔct=Δct gruppo sperimentale-Δct gruppo di controllo
Infine, calcola il multiplo della differenza nel livello di espressione:
Change Fold=2-ΔΔct (corrispondente alla funzione excel è POWER)
Prodotti correlati:
Tempo di pubblicazione: 20 maggio-2023