Le tre lettere SNP sono onnipresenti nello studio della genetica delle popolazioni.Indipendentemente dalla ricerca sulle malattie umane, dal posizionamento dei tratti delle colture, dall'evoluzione degli animali e dall'ecologia molecolare, gli SNP sono necessari come base.Tuttavia, se non si ha una profonda comprensione della genetica moderna basata sul sequenziamento ad alto rendimento e, di fronte a queste tre lettere, sembra "lo sconosciuto più familiare", allora non è possibile effettuare la ricerca successiva.Quindi, prima di intraprendere una ricerca di follow-up, diamo un'occhiata a cos'è l'SNP.
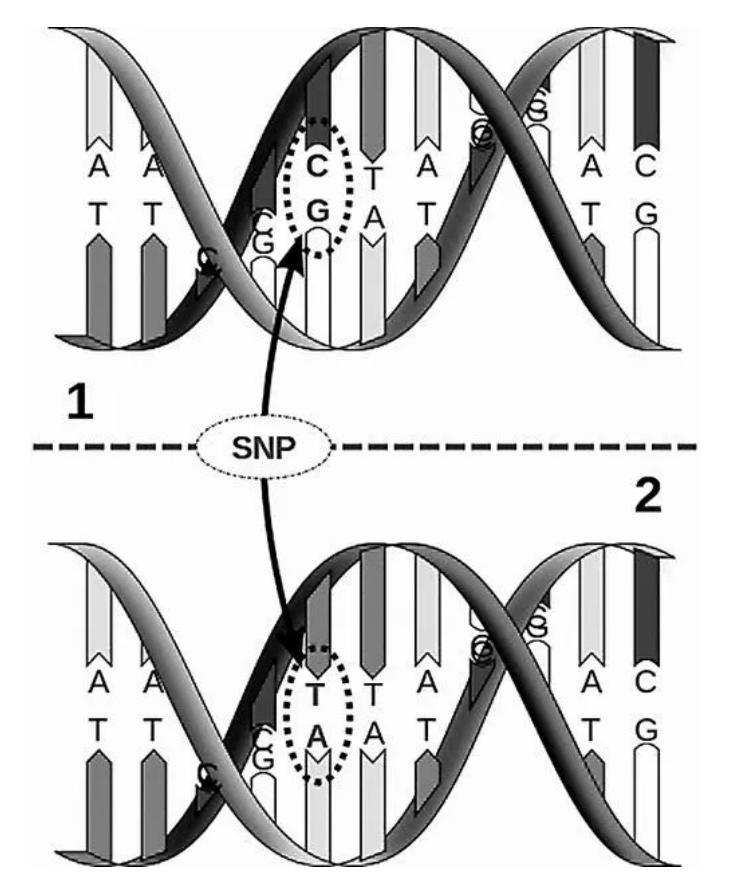
SNP (single nucleotide polymorphism), possiamo vedere dal suo nome inglese completo, si riferisce alla variazione o polimorfismo a singolo nucleotide.Ha anche un nome separato, chiamato SNV (variazione a singolo nucleotide).In alcuni studi sull'uomo, solo quelli con una frequenza di popolazione superiore all'1% sono chiamati SNP, ma in generale i due possono essere mescolati.Quindi possiamo dire che SNP, polimorfismo a singolo nucleotide, si riferisce alla mutazione in cui un nucleotide nel genoma è sostituito da un altro nucleotide.Ad esempio, nella figura seguente, una coppia di basi AT viene sostituita con una coppia di basi GC, che è un sito SNP.
Immagine
Tuttavia, che si tratti di un "polimorfismo a singolo nucleotide" o di una "variazione a singolo nucleotide", è relativamente parlando, quindi i dati SNP richiedono il risequenziamento del genoma come base, ovvero i dati di sequenziamento vengono sequenziati dopo che il genoma dell'individuo è stato sequenziato.Rispetto al genoma, il sito che differisce dal genoma viene rilevato come sito SNP.
In termini di SNP sugli ortaggi,il kit Plant Direct PCRpuò essere utilizzato per il rilevamento rapido.
In termini di tipi di mutazione, SNP include la transizione e la trasversione.La transizione si riferisce alla sostituzione di purine con purine o pirimidine con pirimidine.La trasversione si riferisce alla sostituzione reciproca tra purine e pirimidine.La frequenza di occorrenza sarà diversa e la probabilità di transizione sarà superiore a quella di trasversione.
In termini di dove si verifica l'SNP, diversi SNP avranno effetti diversi sul genoma.Gli SNP che si verificano nella regione intergenica, cioè la regione tra i geni sul genoma, potrebbero non influenzare la funzione del genoma e le mutazioni nell'introne o nella regione del promotore a monte del gene possono avere un certo impatto sul gene;Le mutazioni che si verificano nelle regioni dell'esone dei geni, a seconda che causino cambiamenti negli amminoacidi codificati, hanno effetti diversi sulle funzioni geniche.(Naturalmente, anche se due SNP causano differenze negli amminoacidi, hanno effetti diversi sulla struttura della proteina e, in ultima analisi, gli effetti sul fenotipo biologico possono essere molto diversi).
Tuttavia, il numero di SNP che si verificano nelle posizioni dei geni è solitamente significativamente inferiore a quello delle posizioni non geniche, poiché un SNP che influenza la funzione di un gene di solito ha un impatto negativo sulla sopravvivenza dell'individuo, con il risultato che l'individuo che portava questo SNP nel gruppo Tra di loro è stato eliminato.
Naturalmente, per gli organismi diploidi, i cromosomi esistono in coppia, ma è impossibile che una coppia di cromosomi sia esattamente la stessa per ogni base.Pertanto, alcuni SNP appariranno anche eterozigoti, cioè ci sono due basi in questa posizione sul cromosoma.In un gruppo, i genotipi SNP di individui diversi vengono aggregati insieme, il che diventa la base per la maggior parte delle analisi successive.In combinazione con i tratti, si può giudicare se l'SNP come marcatore molecolare è collegato ai tratti, si può giudicare il QTL (quantitative trait locus) del tratto e si può eseguire il GWAS (studio di associazione sull'intero genoma) o la costruzione della mappa genetica;SNP può essere utilizzato come marcatore molecolare Giudicare la relazione evolutiva tra gli individui;è possibile eseguire lo screening degli SNP funzionali e studiare le mutazioni correlate alla malattia;è possibile utilizzare le variazioni di frequenza degli alleli SNP o i tassi eterozigoti e altri indicatori per determinare le regioni selezionate sul genoma... ecc., combinate con il presente Con lo sviluppo del sequenziamento ad alto rendimento, è possibile ottenere centinaia di migliaia o più siti SNP da una serie di dati di sequenziamento.Si può affermare che SNP è ormai diventato la pietra angolare della ricerca genetica di popolazione.
Naturalmente, i cambiamenti nelle basi nel genoma non sono sempre la sostituzione di una base con un'altra (sebbene questa sia la più comune).È anche possibile che manchino una o poche basi o due basi.Diverse altre basi sono state inserite nel mezzo.Questa piccola gamma di inserzioni e delezioni è chiamata collettivamente InDel (inserimento e delezione), che si riferisce specificamente all'inserimento e alla delezione di brevi frammenti (una o più basi).InDel che si verifica nella posizione di un gene può anche avere un impatto sulla funzione del gene, quindi a volte InDel può anche svolgere un ruolo importante nella ricerca.Ma nel complesso, lo status di SNP come pietra angolare della genetica delle popolazioni è ancora incrollabile.
Tempo di pubblicazione: 27 agosto-2021