Specificità del rilevamento
Nella maggior parte dei casi, lo scopo del disegno del primer è quello di massimizzare la specificità della PCR.Ciò è determinato dall'influenza più o meno prevedibile di molte variabili.Una variabile importante è la sequenza all'estremità 3′ del primer.
È importante sottolineare che i test PCR progettati per la specificità hanno maggiori probabilità di mantenere un'elevata efficienza su un ampio intervallo dinamico, poiché il test non produce prodotti di amplificazione non specifici, competendo quindi con i reagenti PCR o inibendo la reazione di amplificazione principale.
Naturalmente, in alcuni casi, la specificità non è la cosa più importante, ad esempio, quando l'obiettivo è quantificare agenti patogeni strettamente correlati ma diversi, sono richiesti standard di progettazione, ottimizzazione e verifica speciali.
La curva di melting è un metodo standard per valutare la specificità degli ampliconi, almeno in termini di amplificazione di un singolo bersaglio.Tuttavia, va sottolineato che le curve di melting possono essere fuorvianti perché, ad esempio, possono essere influenzate dagli effetti combinati di primer non ottimali e basse concentrazioni di template.
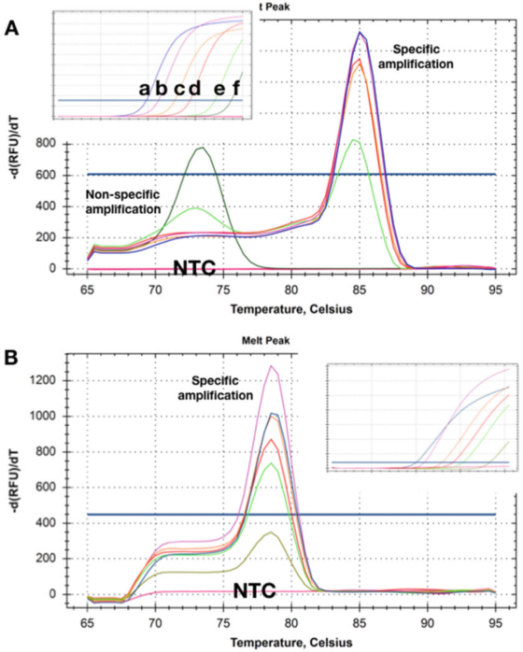
P5 |La curva di fusione mostra gli spostamenti Tm ottenuti da due rilevamenti di diverse quantità di due DNA bersaglio.
A. A concentrazioni più elevate (ad)), non vi è alcun dimero di primer evidente dopo che la misurazione qPCR è stata completata.Quando la concentrazione dello stampo scende a 50 copie (e), inizia ad apparire un prodotto non specifico che diventa l'unico prodotto con la concentrazione più bassa (f).
B. Il test ha registrato la stessa Tms a tutte le concentrazioni target e non vi era alcun dimero di primer evidente anche alla concentrazione più bassa (5 copie).Quando si utilizzano questi due metodi di rilevamento, non sono stati rilevati prodotti di amplificazione negli NTC.
P5 mostra le curve di dissoluzione ottenute con campioni in cui il templato è presente a diverse concentrazioni.P 5a mostra che alle due concentrazioni più basse, i Tms dei prodotti di amplificazione non specifici prodotti sono inferiori a quelli degli ampliconi specifici.
Ovviamente, questo metodo di rilevamento non può essere utilizzato in modo affidabile per rilevare obiettivi che esistono in basse concentrazioni.
È interessante notare che gli NTC, cioè i campioni senza DNA, non hanno registrato prodotti di amplificazione (non specifici), indicando che il DNA genomico di fondo può partecipare all'amplificazione/polimerizzazione non specifica.
A volte non è possibile rimediare a tali primer di fondo e all'amplificazione non specifica, ma è spesso possibile progettare un metodo di rilevamento che non abbia un'amplificazione non specifica in nessuna concentrazione di templato e NTC (P 5b).
Qui, anche la registrazione dell'amplificazione della concentrazione target con un Cq di 35 produrrà una specifica curva di dissoluzione.Allo stesso modo, gli NTC non hanno mostrato segni di amplificazione non specifica.A volte, il comportamento di rilevamento può dipendere dal liquore madre e in determinate composizioni tampone viene rilevata solo un'amplificazione non specifica, che può essere correlata a diverse concentrazioni di Mg2+.
Stabilità del rilevamento
L'ottimizzazione di Ta è un passo utile nel processo di verifica e ottimizzazione empirica del rilevamento qPCR.Fornisce un'indicazione diretta della robustezza del set di primer mostrando la temperatura (o l'intervallo di temperatura) che produce il Cq più basso senza amplificare l'NTC.
La differenza di sensibilità da due a quattro volte potrebbe non essere importante per le persone con un'elevata espressione di mRNA, ma per i test diagnostici potrebbe significare la differenza tra risultati positivi e falsi negativi.
Le proprietà Ta dei primer qPCR possono variare notevolmente.Alcuni test non sono molto robusti e se non vengono eseguiti con il valore Ta ottimale dei primer, collasseranno rapidamente.
Questo è importante perché questo tipo di rilevamento è spesso problematico nel mondo reale e la purezza del campione, la concentrazione del DNA o la presenza di altro DNA potrebbero non essere ottimali.
Inoltre, il numero di copie target può variare in un ampio intervallo e i reagenti, gli utensili di plastica o gli strumenti possono essere diversi da quelli utilizzati durante l'impostazione del test.
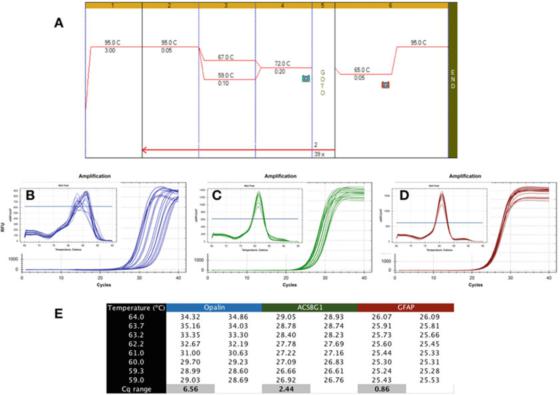
P6|Il gradiente di temperatura mostra la diversa robustezza del rilevamento PCR.
A. Utilizzare il mastermix Sensifast SYBR di Bioline (numero di catalogo BIO-98050) per eseguire la PCR sul cDNA preparato dall'RNA del cervello umano.
B. Utilizzare lo strumento CFX qPCR di Bio-Rad per registrare la mappa di amplificazione e la curva di dissoluzione dell'apalene (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Grafico di amplificazione e curva di fusione di ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Grafico di amplificazione e curva di dissoluzione di GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs registrato a diverse temperature di ricottura, che mostra la differenza di Cq registrata con un gradiente di temperatura di 7°C.
P 6 mostra un risultato tipico di un test indesiderabile, in cui qPCR è stato eseguito utilizzando un gradiente Tas tra 59C e 67C (P 6a), utilizzando primer per tre geni specifici del cervello umano.
Si può vedere dal grafico di amplificazione che i primer Opalin sono tutt'altro che ideali perché il loro intervallo Ta ottimale è molto stretto (Figura 6b), ovvero i Cq sono ampiamente dispersi, con il risultato che i Cq sono significativamente rispetto al loro Cq ottimale Basso.
Questo metodo di rilevamento è instabile e può portare a un'amplificazione subottimale.Pertanto, questa coppia di primer dovrebbe essere riprogettata.Inoltre, l'analisi della curva di fusione (riquadro) mostra che anche la specificità di questo metodo di rilevamento può essere problematica, poiché la curva di fusione di ogni Ta è diversa.
Il metodo di rilevamento ACSBG1 mostrato in P 6c è più robusto del metodo di rilevamento Opalin sopra, ma è ancora lontano dall'ideale ed è probabile che possa essere migliorato.
Tuttavia, sottolineiamo che non esiste una connessione necessaria tra robustezza e specificità, perché la curva di dissoluzione prodotta da questo metodo di rilevamento mostra lo stesso valore di picco in tutti i Tas (riquadro).
D'altra parte, il test di robustezza è molto più tollerante, producendo Cq simili in un'ampia gamma di Tas, come nel test GFAP mostrato in P 6d.
La differenza in Cqs ottenuta nello stesso intervallo di 8 gradi Celsius è inferiore a 1 e la curva di dissoluzione (riquadro) conferma le caratteristiche di rilevamento in questo intervallo di temperatura.Vale la pena notare che la Tas calcolata e l'intervallo Ta effettivo possono essere molto diversi.
Esistono molte linee guida progettate per aiutare i ricercatori a progettare primer efficienti, la maggior parte delle quali si basano su regole consolidate e molta attenzione è stata prestata all'estremità 3′ dei primer.Si consiglia spesso di includere un G o un C all'estremità 3' e due basi G o C (morsetto GC), ma non più di due delle ultime 5 basi.
In pratica, queste regole possono guidare i ricercatori, ma non sono necessariamente corrette in tutte le circostanze.
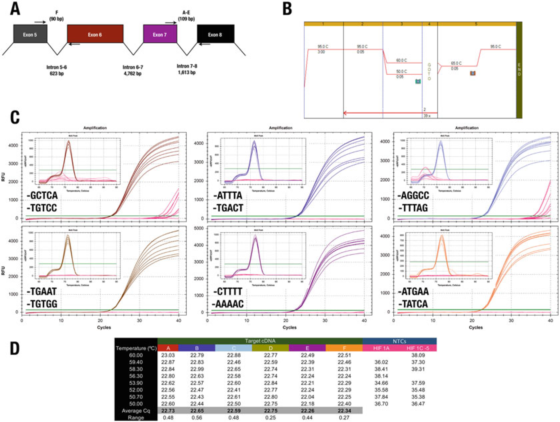
P7 |L'estremità 3′ del primer ha scarso effetto sulla specificità o sull'efficienza.
A. La posizione dei primer per il gene umano HIF-1α (NM_181054.2).
B. Utilizzare il liquido madre Agilent Brilliant III SYBR Green (n. di cat. 600882) per amplificare sei elementi del test.
C. Grafico di amplificazione e curva di fusione registrati dallo strumento CFX qPCR di Bio-Rad e primer 3'end.Gli NTC sono mostrati in rosso.
D. Cqs record di ogni elemento di prova
Ad esempio, il risultato in P 7 contraddice la regola 3′end.Tutti i progetti producono fondamentalmente gli stessi risultati, con solo due combinazioni di primer che portano all'amplificazione non specifica in NTC.
Tuttavia, non possiamo supportare l'effetto della clip GC, perché in questo caso, l'utilizzo di A o T come massimo di 30 basi non riduce la specificità.
Il test C, dove il primer F termina in GGCC, ha registrato Cqs in NTC, indicando che si potrebbe voler evitare queste sequenze all'estremità 30.Sottolineiamo che l'unico modo per determinare la migliore sequenza di estremità 3′ di una coppia di primer è valutare sperimentalmente alcuni primer candidati.
Efficienza di amplificazione
È importante sottolineare che, sebbene il rilevamento PCR non specifico non possa mai diventare specifico, l'efficienza di amplificazione può essere regolata e massimizzata in molti modi diversi modificando l'enzima, il liquore madre, gli additivi e le condizioni del ciclo.
Per valutare l'efficienza del rilevamento PCR, è meglio utilizzare una diluizione seriale di 10 o 5 volte l'acido nucleico target, ovvero il "metodo della curva standard".
Se si utilizzano ampliconi PCR o target di DNA sintetico per generare una curva standard, le diluizioni seriali di questi target devono essere miscelate con una quantità costante di DNA di fondo (come il DNA genomico).
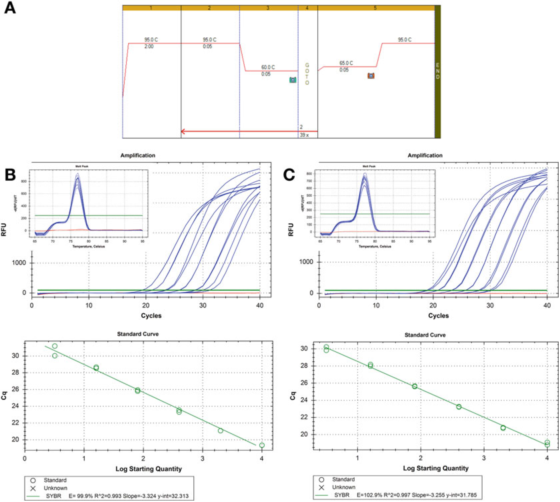
P8 |Curva di diluizione per valutare l'efficienza della PCR.
A. Utilizzare i primer per HIF-1: F: AAGAACTTTTAGGCCGCTCA e R: TGTCCTGTGGTGACTTGTCC e la mastermix Brilliant III SYBR Green di Agilent (numero di catalogo 600882) per la PCR e le condizioni della curva di melting.
B. 100 ng di RNA sono stati retrotrascritti, diluiti 2 volte, e campioni di cDNA diluiti in serie sono stati diluiti 5 volte a 1 ng di DNA genomico umano.La curva di fusione è mostrata nel riquadro.
C. La reazione RT, la diluizione e la diluizione seriale sono state ripetute per il secondo campione di cDNA e i risultati sono stati simili.
P 8 mostra due curve standard, utilizzando lo stesso metodo di rilevamento su due diversi campioni di cDNA, il risultato è la stessa efficienza, circa il 100%, e anche il valore R2 è simile, ovvero il grado di adattamento tra i dati sperimentali e la linea di regressione o il grado di linearità dei dati.
Le due curve standard sono comparabili, ma non esattamente uguali.Se lo scopo è quello di quantificare accuratamente l'obiettivo, va notato che è inaccettabile fornire un calcolo del numero di copie senza spiegare l'incertezza
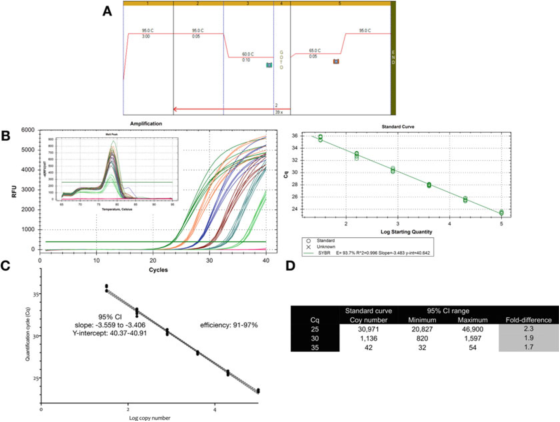
P9 |Incertezza di misura associata alla quantificazione mediante una curva standard.
A. Utilizzare i primer per GAPDH (NM_002046) per eseguire la PCR e le condizioni della curva di fusione.F: ACAGTTGCCATGTAGACC e R: TAACTGGTTGAGCACAGG e mastermix Sensifast SYBR di Bioline (numero di catalogo BIO-98050).
B. Grafico di amplificazione, curva di melting e curva standard registrati con lo strumento CFX qPCR di Bio-Rad.
C. Grafico della curva standard e intervallo di confidenza al 95% (CI).
D. Il numero di copie e l'intervallo di confidenza al 95% dei tre valori Cq derivati dalla curva di diluizione.
P 9 mostra che per un test ottimizzato, la variabilità intrinseca di una singola curva standard è di circa 2 volte (intervallo di confidenza del 95%, da minimo a massimo), che può essere la più piccola variabilità che ci si può aspettare.
Prodotti correlati:
Kit per PCR diretta a coda di topo
Kit per PCR diretta su tessuto animale
Tempo di pubblicazione: 30 settembre 2021