È noto che nel dogma centrale l'RNA è il mediatore trascrizionale tra il DNA e l'espressione proteica.Rispetto al rilevamento del DNA, il rilevamento dell'RNA può riflettere in modo più obiettivo l'espressione genica negli organismi.Gli esperimenti che coinvolgono l'RNA includono: qRT-PCR, RNA-Seq e rilevamento del gene di fusione, ecc. Sulla base delle caratteristiche dell'RNA stesso (l'anello di zucchero dell'RNA ha un gruppo idrossile libero in più rispetto all'anello di zucchero del DNA), insieme a un gran numero di RNasi nell'ambiente, l'RNA è più instabile e più facile da degradare rispetto al DNA.Spazzatura dentro, spazzatura fuori, se la qualità dell'RNA non è buona, allora i risultati sperimentali devono essere insoddisfacenti, manifestati specificamente come dati imprecisi o scarsa ripetibilità.Pertanto, si dovrebbe prestare maggiore attenzione all'elaborazione dell'RNA e anche il collegamento del controllo di qualità è più importante per garantire la precisione e l'accuratezza dei successivi dati sperimentali.
Per il controllo di qualità dell'RNA, ci sono generalmente i seguenti metodi comunemente usati:
- Spettrofotometria
- elettroforesi su gel di agarosio
- Bioanalizzatore Agilent
- PCR quantitativa fluorescente in tempo reale
- Metodo di tintura fluorescente Qubit
01 Spettrofotometria
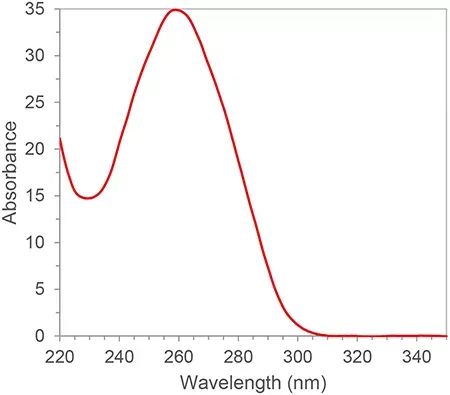
L'RNA ha doppi legami coniugati e ha un picco di assorbimento a una lunghezza d'onda di 260 nm.Secondo la legge di Lambert-Beer, possiamo calcolare la concentrazione di RNA dal picco di assorbimento a 260 nm.Inoltre, possiamo anche calcolare la purezza dell'RNA in base al rapporto tra i picchi di assorbimento a 260 nm, 280 nm e 230 nm.280 nm e 230 nm sono rispettivamente i picchi di assorbimento delle proteine e delle piccole molecole.Il rapporto tra A260/A280 e A260/A230 della purezza dell'RNA qualificato deve essere maggiore di 2. Se è inferiore a 2, significa che c'è una contaminazione da proteine o piccole molecole nel campione di RNA e deve essere nuovamente purificato.Le fonti di contaminazione influenzeranno gli esperimenti a valle, come l'inibizione dell'efficienza di amplificazione delle reazioni PCR, con conseguenti risultati quantitativi imprecisi.La purezza dell'RNA ha una grande influenza sui risultati successivi, quindi la spettrofotometria è generalmente un collegamento indispensabile per il controllo della qualità nella prima fase degli esperimenti con l'acido nucleico.
Figura 1. Tipico spettro di assorbimento di RNA/DNA
02 Elettroforesi su gel di agarosio
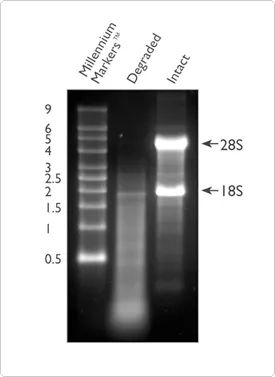
Oltre alla purezza, l'integrità dell'RNA è anche uno degli indicatori importanti per giudicare la qualità dell'RNA.La degradazione dell'RNA porterà a un gran numero di brevi frammenti nel campione, quindi il numero di frammenti di RNA che possono essere effettivamente rilevati e coperti dalla sequenza di riferimento sarà ridotto.L'integrità dell'RNA può essere verificata mediante elettroforesi dell'RNA totale su gel di agarosio all'1%.Questo metodo può configurare autonomamente il gel o utilizzare il sistema E-Gel™ prefabbricato per i test di integrità.Più dell'80% dell'RNA totale è RNA ribosomiale, la maggior parte del quale è composta da 28S e 18S rRNA (nei sistemi dei mammiferi).L'RNA di buona qualità mostrerà due evidenti barre luminose, che sono barre luminose 28S e 18S, rispettivamente, a 5 Kb e 2 Kb, e il rapporto tenderà ad essere vicino a 2:1.Se è in uno stato diffuso, significa che il campione di RNA potrebbe essere stato degradato e si consiglia di utilizzare il metodo descritto in seguito per testare ulteriormente la qualità dell'RNA.
Figura 2. Confronto tra RNA degradato (corsia 2) e intatto (corsia 3) su elettroforesi su gel di agarosio
03 Bioanalizzatore Agilent
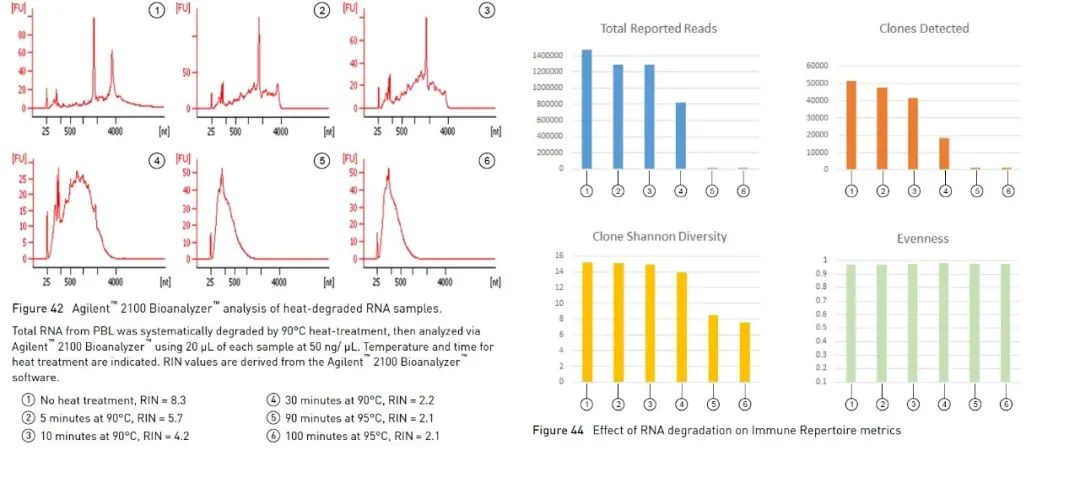
Oltre al metodo di elettroforesi su gel di agarosio sopra descritto, che può aiutarci a identificare l'integrità dell'RNA in modo semplice e rapido, possiamo anche utilizzare il bioanalizzatore Agilent per determinare l'integrità dell'RNA.Utilizza una combinazione di microfluidica, elettroforesi capillare e fluorescenza per valutare la concentrazione e l'integrità dell'RNA.Utilizzando l'algoritmo integrato per analizzare il profilo del campione di RNA, il bioanalizzatore Agilent è in grado di calcolare un valore di integrità dell'RNA di riferimento, il numero di integrità dell'RNA (di seguito denominato RIN) [1].Maggiore è il valore di RIN, maggiore è l'integrità dell'RNA (1 è estremamente degradato, 10 è il più completo).Alcuni esperimenti che coinvolgono l'RNA suggeriscono di utilizzare RIN come parametro per la valutazione della qualità.Prendendo come esempio gli esperimenti di sequenziamento ad alto rendimento (di seguito denominati NGS), le linee guida di Oncomine™ Human Immune Repertoire, utilizzate per rilevare i recettori dell'antigene delle cellule B e T nella serie di pannelli Oncomine di Thermo Fisher, suggeriscono che è possibile misurare campioni con valori RIN superiori a 4, letture e cloni più efficaci (Figura 3).Esistono diversi intervalli consigliati per diversi panel e spesso un RIN più elevato può fornire dati più efficaci.
Figura 3, negli esperimenti Oncomine™ Human Immune Repertoire, i campioni con RIN maggiore di 4 possono rilevare letture più efficaci e cloni di cellule T.【2】
Tuttavia, anche il valore RIN presenta alcune limitazioni.Sebbene il RIN abbia un'elevata correlazione con la qualità dei dati sperimentali NGS, non è adatto per i campioni FFPE.I campioni FFPE sono stati trattati chimicamente per molto tempo e l'RNA estratto ha generalmente un valore RIN relativamente basso.Tuttavia, ciò non significa che i dati effettivi dell'esperimento debbano essere insoddisfacenti.Per valutare con precisione la qualità dei campioni FFPE, dobbiamo utilizzare misurazioni diverse dal RIN.Oltre al RIN, il bioanalizzatore Agilent può anche calcolare il valore DV200 come parametro di valutazione della qualità dell'RNA.DV200 è un parametro che calcola la proporzione di frammenti superiori a 200 bp in un campione di RNA.DV200 è un indicatore migliore della qualità del campione FFPE rispetto a RIN.Per l'RNA estratto da FFPE, ha una correlazione molto elevata con il numero di geni che possono essere effettivamente rilevati e la diversità dei geni [3].Sebbene DV200 possa compensare le carenze nel rilevamento della qualità di FFPE, il bioanalizzatore Agilent non è ancora in grado di analizzare in modo completo i problemi di qualità nei campioni di RNA, inclusa la presenza di inibitori nei campioni.Gli stessi inibitori possono influenzare l'efficienza di amplificazione degli esperimenti a valle e ridurre la quantità di dati utili.Per sapere se c'è un inibitore nel campione, possiamo adottare il metodo PCR quantitativo fluorescente in tempo reale descritto di seguito.
04 PCR quantitativa fluorescente in tempo reale
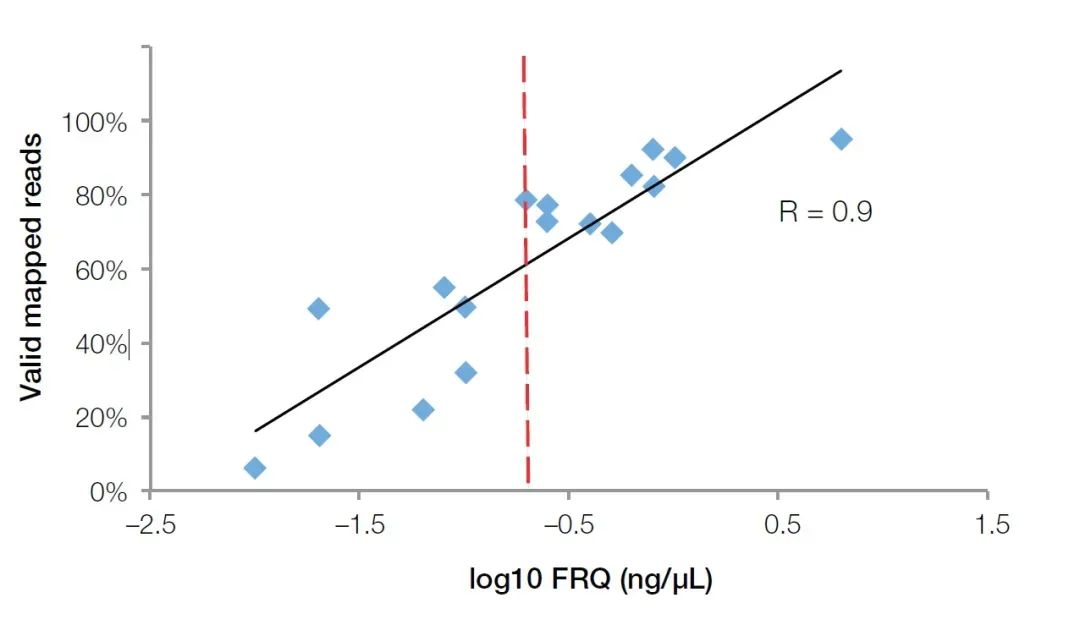
Il metodo PCR quantitativo fluorescente in tempo reale può non solo rilevare gli inibitori nel campione, ma anche riflettere accuratamente la qualità dell'RNA nel campione FFPE.Rispetto agli analizzatori biologici Agilent, gli strumenti quantitativi di fluorescenza in tempo reale sono più popolari nei principali laboratori biologici grazie alla loro più ampia applicazione.Per testare la qualità dei campioni di RNA, dobbiamo solo acquistare o preparare sonde primer per i geni di riferimento interni, come GUSB (Cat n. Hs00939627).Utilizzando questo set di primer, sonde e standard (RNA totale di concentrazione nota) per condurre esperimenti quantitativi assoluti, la concentrazione effettiva del frammento di RNA può essere calcolata come standard di valutazione della qualità dell'RNA (quantificazione funzionale dell'RNA (FRQ) in breve).In un test NGS, abbiamo scoperto che il FRQ dei campioni di RNA ha una correlazione molto elevata con il volume di dati effettivo.Per tutti i campioni superiori a 0,2 ng/uL FRQ, almeno il 70% delle letture può effettivamente coprire la sequenza di riferimento (Figura 4).
Figura 4, il valore FRQ rilevato dal metodo quantitativo di fluorescenza ha una correlazione molto elevata (R2>0,9) con i dati effettivi ottenuti nell'esperimento NGS.La linea rossa è il valore FRQ pari a 0.2 ng/uL (log10 = -0.7).【4】
Oltre ad essere applicabile ai campioni FFPE, il metodo PCR quantitativo in tempo reale può anche monitorare efficacemente gli inibitori nei campioni.Possiamo aggiungere il campione da rilevare nel sistema di reazione con il controllo positivo interno (IPC) e il relativo dosaggio, quindi eseguire la quantificazione della fluorescenza per ottenere il valore Ct.Se il valore Ct è in ritardo rispetto al valore Ct nella reazione senza campione, indica che l'inibitore è presente nel campione e inibisce l'efficienza di amplificazione nella reazione.
05 Metodo colorante fluorescente Qubit
Il fluorometro Qubit è il piccolo dispositivo più comunemente usato per il rilevamento della concentrazione e della purezza dell'acido nucleico, che è facile da usare ed esiste in quasi tutti i laboratori di biologia molecolare.Calcola accuratamente la concentrazione di acido nucleico rilevando e colorante fluorescente legante l'acido nucleico (reagente di rilevamento Qubit).Qubit ha un'elevata sensibilità e specificità e può quantificare accuratamente l'RNA fino alla concentrazione di pg/µL.Oltre alla ben nota capacità di quantificare con precisione la concentrazione di acido nucleico, l'ultimo nuovo modello di Thermo Fisher, Qubit 4.0, può anche rilevare l'integrità dell'RNA.Il sistema di rilevamento dell'RNA di Qubit 4.0 (RNA IQ Assay) rileva l'integrità dell'RNA rilevando simultaneamente due specifici coloranti fluorescenti.Questi due coloranti fluorescenti possono legarsi rispettivamente a grandi frammenti e piccoli frammenti di RNA.Questi due coloranti fluorescenti indicano la proporzione di grandi frammenti di RNA nel campione e da questo è possibile calcolare il valore IQ (integrità e qualità) che rappresenta la qualità dell'RNA.Il valore IQ è applicabile a entrambi i campioni FFPE e non FFPE e ha una grande influenza sulla successiva qualità del sequenziamento.Prendendo come esempio gli esperimenti NGS, negli esperimenti di test RNA-Seq eseguiti sulla piattaforma Ion torrent™, la maggior parte dei campioni con valori di QI superiori a 4 presentava almeno il 50% di letture effettive (Figura 5).Rispetto ai metodi di rilevamento sopra menzionati, Qubit IQ Assay non solo è più conveniente da usare e richiede meno tempo (entro cinque minuti), ma ha anche un'ottima correlazione tra il valore del QI del parametro misurato e la qualità dei dati degli esperimenti a valle.
Figura 5, c'è una grande correlazione tra il valore QI RNA Qubit e le letture mappate di RNA-Seq.【5】
Attraverso l'introduzione di cui sopra, credo che tutti abbiano una comprensione sufficiente dei diversi metodi di controllo della qualità dell'RNA.In pratica puoi scegliere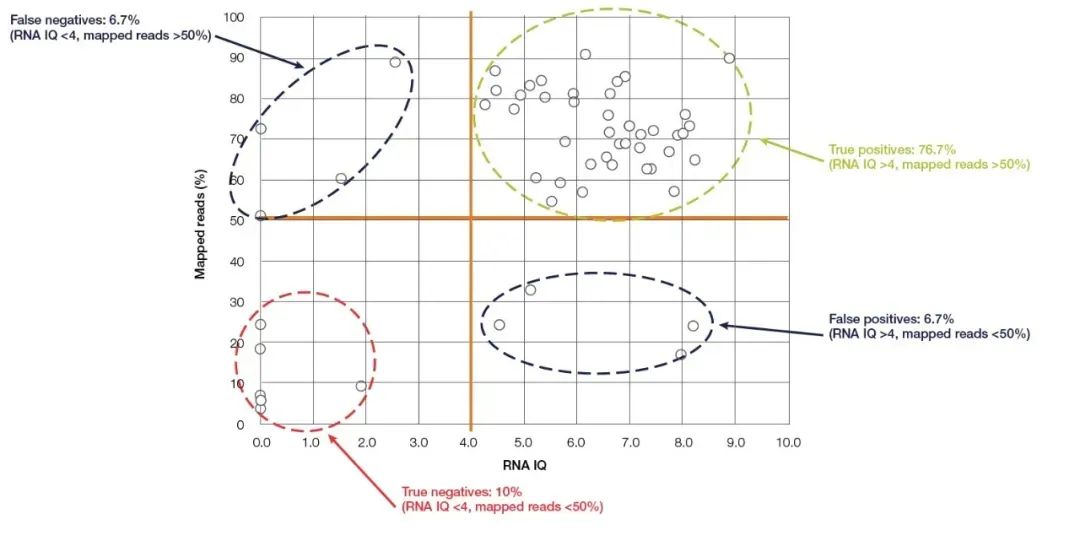 il metodo corrispondente in base al tipo di campione e agli strumenti esistenti.Solo controllando bene la qualità dell'RNA possiamo evitare il fallimento degli esperimenti successivi causato da una scarsa qualità del campione, risparmiando così tempo prezioso, energia e costi.
il metodo corrispondente in base al tipo di campione e agli strumenti esistenti.Solo controllando bene la qualità dell'RNA possiamo evitare il fallimento degli esperimenti successivi causato da una scarsa qualità del campione, risparmiando così tempo prezioso, energia e costi.
Prodotti di riferimento:
Kit di isolamento dell'RNA totale animale
Kit di isolamento dell'RNA totale delle cellule
Riferimenti
【1】 Schroeder, A., Mueller, O., Stocker, S. et al.Il RIN: un numero di integrità dell'RNA per l'assegnazione dei valori di integrità alle misurazioni dell'RNA.BMC Molecular Biol 7, 3 (2006).https:// doi.org/10.1186/1471-21 99-7-3
【2】Manuale utente Oncomine Human Immune Repertoire (Pub. N. MAN0017438 Rev. C.0).
【3】Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, Metriche di qualità avanzate per la valutazione dell'RNA derivato da campioni di tessuto inclusi in paraffina fissati in formalina, Scienze tossicologiche, Volume 170, Numero 2, agosto 2019, Pagine 357–373,https://doi.org/10.1093/toxsci/
Tempo di pubblicazione: giu-12-2023