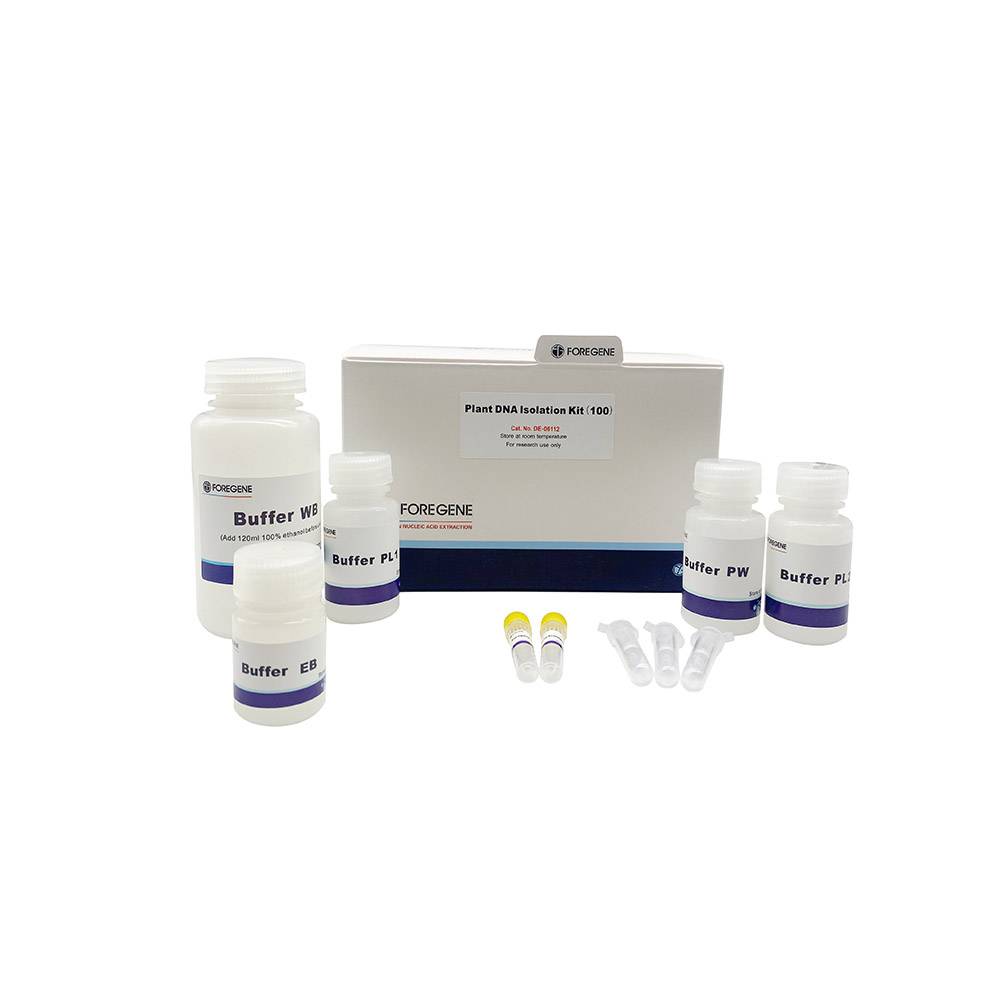
Fonte di fabbrica High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix
Sottolineiamo il miglioramento e introduciamo nuove soluzioni sul mercato quasi ogni anno per la fonte di fabbrica High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix, ci dedichiamo a fornire tecnologia e opzioni di purificazione qualificate per te personalmente!
Sottolineiamo il miglioramento e introduciamo nuove soluzioni nel mercato quasi ogni anno perChina PCR Kit e PCR, Fino ad ora, l'elenco delle merci è stato aggiornato regolarmente e ha attirato clienti da tutto il mondo.I fatti approfonditi sono spesso ottenuti nel nostro sito Web e ti verrà servito con un servizio di consulenza di alta qualità dal nostro gruppo post-vendita.Ti aiuteranno a ottenere un riconoscimento approfondito dei nostri prodotti e a concludere una trattativa soddisfatta.Anche l'azienda che va alla nostra fabbrica in Brasile è la benvenuta in qualsiasi momento.Spero di ottenere le vostre richieste per qualsiasi felice collaborazione.
Manuale di istruzioni:
Sottolineiamo il miglioramento e introduciamo nuove soluzioni sul mercato quasi ogni anno per la fonte di fabbrica High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix, ci dedichiamo a fornire tecnologia e opzioni di purificazione qualificate per te personalmente!
Fonte di fabbricaChina PCR Kit e PCR, Fino ad ora, l'elenco delle merci è stato aggiornato regolarmente e ha attirato clienti da tutto il mondo.I fatti approfonditi sono spesso ottenuti nel nostro sito Web e ti verrà servito con un servizio di consulenza di alta qualità dal nostro gruppo post-vendita.Ti aiuteranno a ottenere un riconoscimento approfondito dei nostri prodotti e a concludere una trattativa soddisfatta.Anche l'azienda che va alla nostra fabbrica in Brasile è la benvenuta in qualsiasi momento.Spero di ottenere le vostre richieste per qualsiasi felice collaborazione.
Guida all'analisi dei problemi
The following is an analysis of the problems that may be encountered in the extraction of plant genomic DNA, hoping to be helpful to your experiments. In addition, for other experimental or technical problems other than operation instructions and problem analysis, we have dedicated technical support to help you. If you have any needs, please contact us: 028-83360257 or E-mali: Tech@foregene.com.
Bassa resa o assenza di DNA
Di solito ci sono molti fattori che influenzano la resa del DNA genomico, inclusa la fonte del campione, l'età del campione, le condizioni di conservazione del campione e l'operazione.
Non è stato possibile ottenere il DNA genomico durante l'estrazione
1. I campioni di tessuto vengono conservati in modo improprio o conservati troppo a lungo, con conseguente degradazione del DNA genomico.
Raccomandazione: conservare i campioni di tessuto in azoto liquido o -20°C;provare a utilizzare campioni appena raccolti per l'estrazione del DNA genomico.
2. Una quantità di campione insufficiente può impedire l'estrazione del DNA genomico corrispondente.
Suggerimento: per i campioni di tessuto che sono stati conservati per lungo tempo o che presentano una grave degradazione del DNA genomico, la quantità di campioni di tessuto può essere opportunamente aumentata per estrarre una quantità considerevole di DNA genomico.La quantità del campione può essere determinata in base alle esigenze del DNA, ma il campione fresco non deve superare i 100 mg e il campione secco non deve superare i 30 mg.
3. Il campione non viene macinato con azoto liquido o posizionato troppo a lungo dopo l'azoto liquido.
Suggerimento: durante l'estrazione del DNA, il campione deve essere completamente macinato con azoto liquido per rompere la parete cellulare;dopo la macinazione, trasferire la polvere campione in PL1 preriscaldato a 65°C appena possibile (una volta sciolta la polvere macinata, il DNA genomico inizierà a degradarsi rapidamente) .
4. La conservazione impropria di Foregene Protease comporta un'attività ridotta o inattivata.
Raccomandazione: confermare le condizioni di conservazione di Foregene Protease o sostituirlo con un nuovo Foregene Protease per l'idrolisi enzimatica.
5. Il kit viene conservato in modo improprio o conservato troppo a lungo, provocando il malfunzionamento di alcuni componenti del kit.
Raccomandazione: acquistare un nuovo kit di estrazione del DNA genomico vegetale per operazioni correlate.
6. Uso improprio del kit.
Suggerimento: acquistare un kit di isolamento del DNA vegetale dedicato ai campioni per l'estrazione e la purificazione del DNA genomico vegetale.
7. Buffer WB senza aggiungere aanidro etanolo.
Raccomandazione: assicurarsi di aggiungere il volume corretto di etanolo assoluto al tampone WB.
8. L'eluente non è stato gocciolato correttamente sulla membrana di silice.
Suggerimento: aggiungere l'eluente preriscaldato a 65℃goccia a goccia al centro della membrana di gel di silice e lasciarla a temperatura ambiente per 5 minuti per aumentare l'efficienza di eluizione.
Estrazione per ottenere DNA genomico a basso rendimento
1. Il campione viene conservato in modo improprio o conservato troppo a lungo, con conseguente degradazione del DNA genomico.
Raccomandazione: conservare i campioni di tessuto a -20℃;provare a utilizzare campioni di tessuto appena raccolti per l'estrazione del DNA genomico.
2. Se la quantità di campioni di tessuto è troppo piccola, il DNA genomico estratto sarà inferiore.
Suggerimento: alcuni campioni di piante sono ricchi di acqua, come piante acquatiche come alghe, ecc., il dosaggio può essere opportunamente aumentato o l'acqua può essere disidratata un po' prima dell'operazione.
3. I campioni non sono stati accuratamente macinati con azoto liquido o sono stati lasciati a temperatura ambiente troppo a lungo dopo la macinazione.
Suggerimento: la macinazione dell'azoto liquido deve essere sufficiente e la parete della cella del campione deve essere rotta il più possibile;subito dopo la macinazione, la polvere del campione dovrebbe essere trasferita a 65℃Buffer PL1 preriscaldato per il passaggio successivo.
4. Non utilizzare il kit corretto.
Raccomandazione: utilizzare un kit di isolamento del DNA vegetale dedicato per estrarre e purificare il DNA genomico vegetale.
5. La conservazione impropria di Foregene Protease determina un'attività ridotta o inattivata.
Raccomandazione: confermare le condizioni di conservazione di Foregene Protease o sostituirlo con un nuovo Foregene Protease per l'idrolisi enzimatica.
6. Problema dell'eluente
Raccomandazione: utilizzare il tampone EB per l'eluizione;se si utilizza ddH2O o altri eluenti, assicurarsi che il pH dell'eluente sia compreso tra 7,0 e 8,5.
7. L'eluente non è gocciolato correttamente
Suggerimento: aggiungere la goccia di eluizione al centro della membrana di silice e lasciarla a temperatura ambiente per 5 minuti per aumentare l'efficienza di eluizione.
8. Il volume dell'eluente è troppo piccolo
Suggerimento: utilizzare l'eluente per l'eluizione del DNA genomico secondo le istruzioni, almeno non meno di 100microlitri.
DNA genomico estratto con bassa purezza
La bassa purezza del DNA genomico porterà al fallimento o allo scarso effetto degli esperimenti a valle, come ad esempio: l'enzima non può essere tagliato e il frammento del gene bersaglio non può essere ottenuto mediante PCR.
1. Varie contaminazioni proteiche, contaminazione da RNA.
Analisi: il tampone PW non è stato utilizzato per lavare la colonna;Il tampone PW non è stato utilizzato per lavare la colonna alla corretta velocità di centrifugazione.
Suggerimento: cercare di garantire che non ci sia precipitazione nel surnatante quando il surnatante viene fatto passare attraverso la colonna;assicurarsi di lavare la colonna di purificazione con Buffer PW secondo le istruzioni e questo passaggio non può essere omesso.
2. Inquinamento da ioni di impurità.
Analisi: la colonna di lavaggio Buffer WB è stata omessa o lavata solo una volta, con conseguente contaminazione ionica residua.
Raccomandazione: assicurarsi di lavare due volte con Buffer WB secondo le istruzioni per rimuovere il più possibile gli ioni residui.
3. Contaminazione da RNasi.
Analisi: l'RNasi esogena viene aggiunta al tampone;un'errata operazione di lavaggio nel tampone PW risulterà in RNasi residua e influirà sulle operazioni sperimentali dell'RNA a valle, come la trascrizione in vitro.
Suggerimento: i kit di estrazione dell'acido nucleico della serie Foregene possono rimuovere l'RNA senza RNasi aggiuntiva e tutti i reagenti nel kit di isolamento del DNA vegetale non necessitano di RNasi;assicurarsi di lavare la colonna di purificazione con Buffer PW secondo le istruzioni e questo passaggio non può essere omesso.
4. Residui di etanolo.
Analisi: dopo aver lavato la colonna di purificazione con Buffer WB, non è stata eseguita alcuna centrifugazione con tubo vuoto.
Raccomandazione: seguire le istruzioni per una corretta centrifugazione con provette vuote.
Manuale di istruzioni:
Manuale di istruzioni del kit di isolamento del DNA vegetale